En utilisant une version améliorée d’AlphaFold, un outil d’IA de prédiction de structures protéiniques développé par Google DeepMind, des chercheurs ont identifié un complexe de trois protéines clés essentiel à la reconnaissance ovule-spermatozoïde. Ce trimère serait commun à tous les vertébrés et son absence entraîne inévitablement l’échec de la fécondation. Cette découverte contredit l’hypothèse de longue date selon laquelle deux protéines, situées respectivement sur les deux gamètes mâle et femelle, suffisent à assurer la fécondation.
La vie de tout organisme sexué débute par la fécondation, notamment la fusion d’un spermatozoïde avec un ovule. Le gamète mâle se déplace vers l’ovule en étant guidé par des signaux chimiques, puis se lie à la surface de celui-ci par le biais d’interactions protéiques spécifiques. Cette interaction permet à leurs membranes de fusionner, enclenchant ensuite la fusion de leurs matériels génétiques pour former un zygote, la cellule unique qui se développe en embryon.
Cependant, malgré les avancées en matière d’imagerie, les mécanismes moléculaires régissant cette interaction demeurent en partie mystérieux. En effet, contrairement à la plupart des cellules des organismes sexués qui évitent la fusion et conservent une identité génétique distincte, le spermatozoïde et l’ovule sont spécialisés dans la fusion. Cela implique une séquence d’événements moléculaires précis et hautement spécifiques, se déroulant principalement au niveau de leurs membranes lipidiques. Ces processus sont à la fois discrets et fugaces, ce qui les rend difficiles à étudier avec les techniques biochimiques standards.
D’autre part, le prélèvement et la conservation d’ovules et de spermatozoïdes viables chez certains animaux de laboratoire, tels que les souris, sont particulièrement complexes. La plupart des études sur la biologie de la reproduction se sont donc principalement concentrées sur les invertébrés marins (comme les oursins), car ils peuvent libérer de grandes quantités d’ovules et de sperme viable dans l’eau.
Afin de surmonter ces défis, les chercheurs de la nouvelle étude ont focalisé leurs travaux sur le poisson-zèbre (Danio rerio), un vertébré qui libère ses ovules (ou œufs) et ses spermatozoïdes dans l’eau. Ils se sont également appuyés sur la puissance de prédiction d’AlphaFold, qui a valu le prix Nobel de chimie de cette année à deux de ses développeurs, afin de déterminer à l’avance les interactions possibles entre les protéines membranaires des gamètes.
L’IA a permis de mettre au jour une protéine essentielle jusqu’ici non identifiée. « Ce n’est plus le vieux concept d’avoir une clé et une serrure pour ouvrir la porte, c’est plus compliqué », explique à la revue Nature Enrica Bianchi de l’Université de Rome Tor Vergata, qui n’a pas participé à l’étude.
Une protéine spermatique supplémentaire identifiée
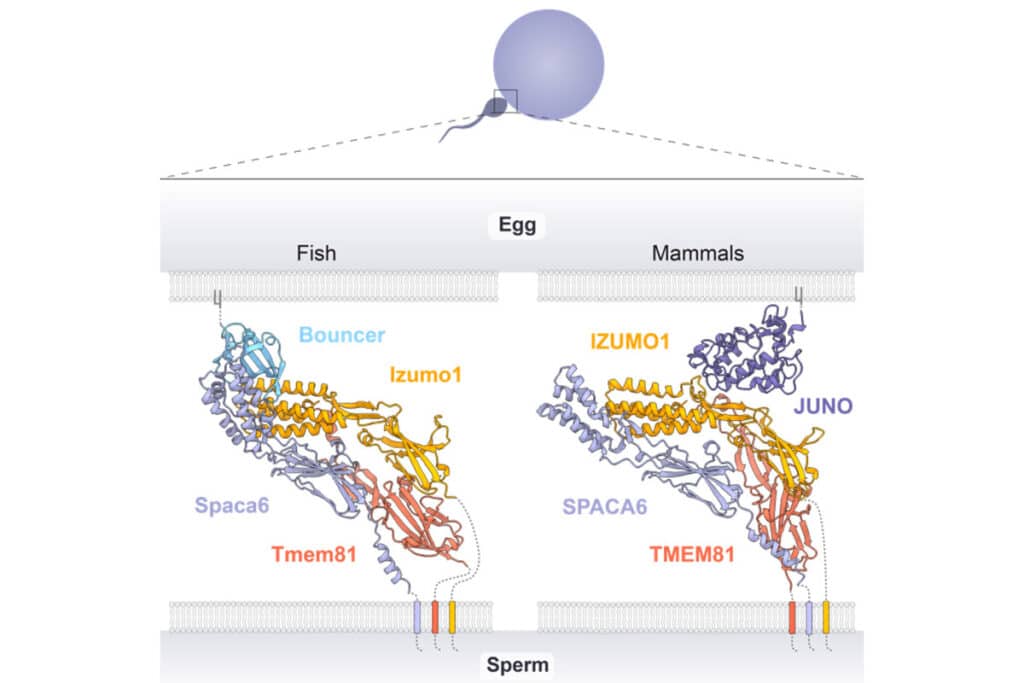
Au cours des 20 dernières années, le criblage génétique a permis d’identifier plusieurs protéines essentielles à l’interaction entre les gamètes mâle et femelle des organismes sexués. Izumo1 est présent au niveau de la membrane du spermatozoïde et se lie à Juno, qui, elle, est présente à la surface de l’ovule, lors de la fécondation. D’autres protéines structurellement similaires à Izumo1 (Spaca6 et Tmem95) sont présentes au niveau de la membrane spermatique, mais ne semblent pas se lier à Juno, selon de précédentes expériences en laboratoire.
En concentrant leur analyse sur les protéines de liaison spermatiques, l’équipe de la nouvelle étude a utilisé AlphaFold Multimer, une version améliorée d’AlphaFold, afin d’identifier d’autres protéines de liaison potentielles. « Nous avons rassemblé une liste de protéines dont la présence est prévue à la membrane du sperme et effectué un criblage bioinformatique comprenant des milliers de prédictions à l’aide d’AlphaFold Multimer », explique dans un communiqué du Research Institute of Molecular Pathology (IMP), Victoria Deneke, coauteure principale de l’étude.
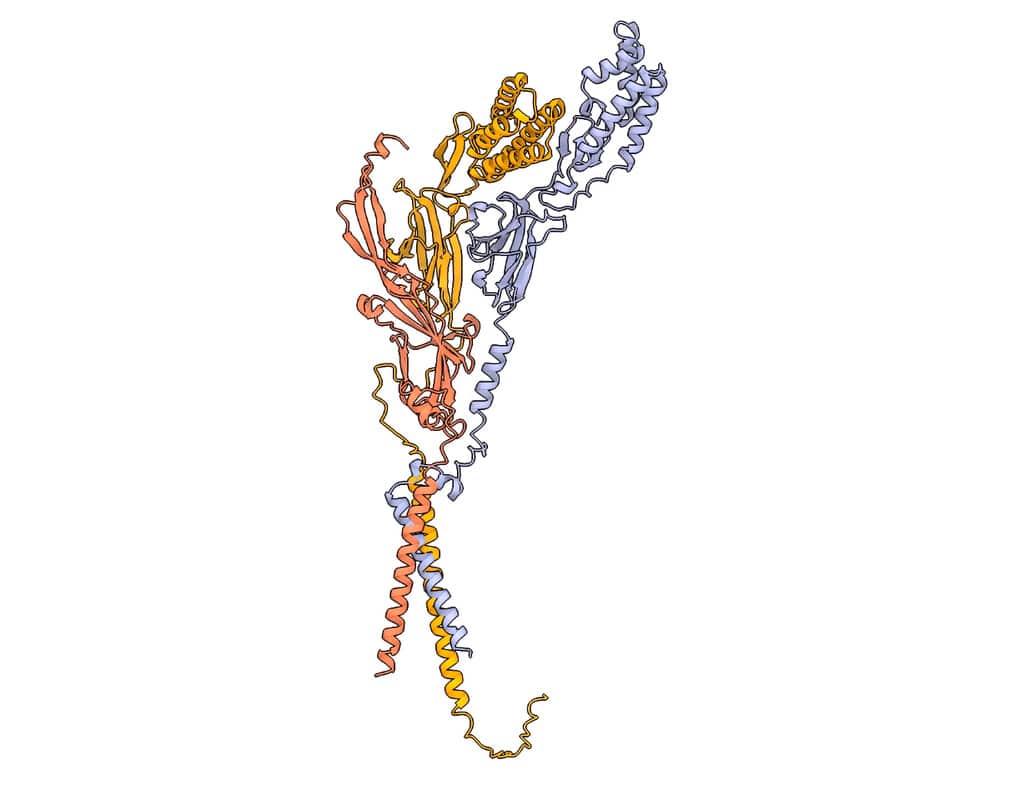
L’algorithme a identifié Tmem81, une protéine supplémentaire jusqu’ici inconnue qui serait essentielle à la liaison avec Juno. Plus précisément, Izumo1 et Spaca6 interagissent non seulement entre elles, mais également avec Tmem81 avant de pouvoir se lier à la protéine ovulaire. Ces premiers résultats démontrent l’importance des algorithmes de prédiction et de modélisation en cas de possibilités de démonstration empiriques limitées.
Un complexe conservé au cours de l’évolution des vertébrés
Afin de tester ces prédictions, les chercheurs ont observé les processus moléculaires de fécondation en premier lieu chez le poisson-zèbre, puis chez la souris et sur des cellules humaines isolées. Ils ont constaté que le trimère est effectivement essentiel à la fécondation chez l’ensemble des modèles.
Le complexe de trois protéines est ancré à la membrane spermatique, tandis que deux d’entre elles constituent un site de liaison permettant au spermatozoïde de s’arrimer à l’ovule via Juno pour les mammifères, et via une protéine équivalente appelée Bouncer chez les poissons. En d’autres termes, Juno et Bouncer servent de verrou d’accès à l’ovule et ne permettent cet accès qu’avec la bonne « clé ». En outre, sans l’interaction du trimère, le gamète mâle est stérile et la fécondation échoue.


Ces résultats suggèrent que contrairement aux protéines ovulaires, le complexe spermatique Izumo1-Spaca6-Tmem81 est commun à tous les vertébrés et a été conservé tout au long de leur évolution. Juno provient par exemple de la duplication d’un récepteur fœtal spécifique aux mammifères et n’est donc présent que chez ces derniers. « Le fait qu’il ait été maintenu au cours de millions d’années d’évolution montre à quel point ce processus de verrouillage et de clé est important, mais ce qui est vraiment surprenant, c’est que le trimère conservé du spermatozoïde utilise des protéines de l’ovule sans lien avec l’évolution pour s’arrimer à la surface de l’ovule », explique Andrea Pauli de l’IMP, également coauteur de l’étude.


Cette découverte pourrait ouvrir la voie à de nouvelles techniques de dépistage de maladies liées à la fertilité. La compréhension de cette interaction moléculaire pourrait aussi potentiellement contribuer au développement de nouvelles interventions thérapeutiques pour l’infertilité. Les résultats de l’étude sont publiés dans la revue Cell.
Vidéo de présentation de l’étude :