Une nouvelle technique d’encodage permet « d’imprimer » et de stocker des données sur de l’ADN de façon similaire à une ancienne presse d’imprimerie. Un système de marquage épigénétique permet d’écrire les données en code binaire, tandis qu’un dispositif de séquençage à nanopores permet leur lecture en sortie. Bien qu’a priori complexe étant donnée sa conception, la technique ne nécessiterait pas de compétence particulière en techniques de laboratoire.
Malgré les avancées actuelles en matière de stockage de données, ces dernières sont loin d’égaler l’ADN. Concentrant d’énormes quantités d’informations dans des volumes minuscules, on estime qu’un seul centimètre cube d’ADN peut stocker l’équivalent de 10 exaoctets (Eo) de données pendant des milliers d’années, voire des millions dans les meilleures conditions. En raison de cet immense potentiel, l’ADN est actuellement exploré en tant que nouveau support de stockage d’informations numériques.
Les techniques actuelles consistent à convertir les données en code binaire en séquences nucléotidiques (adénine, guanine, cytosine et thymine). L’écriture s’effectue par le biais d’une synthèse de brin d’ADN à partir de zéro, c’est-à-dire en ajoutant un à un les bases nucléotidiques selon un ordre prédéterminé. Ces techniques sont cependant limitées en matière de vitesse d’écriture et de longueur de brins synthétisés.
Afin de surmonter ces limites, des approches alternatives, telles que l’écriture parallèle, ont été proposées. Cependant, l’instabilité structurelle et le débit limité de traitement d’information ont entravé leur application. En revanche, l’épigénome peut coder des informations modifiables stables au-dessus de la séquence génomique qui le transporte. L’étude codirigée par l’Université de Pékin propose une technique inspirée des anciennes techniques d’imprimerie et basée sur la transmission des traits épigénomiques.
« Inspirés par l’hérédité épigénomique naturelle et par l’auto-assemblage synthétique de l’ADN, nous avons développé un cadre d’écriture de données ADN non conventionnel qui permet d’écrire de manière stable des bits d’information épigénétique arbitraires (épi-bits) de manière parallèle sur des modèles d’ADN basés sur la méthylation enzymatique guidée par l’auto-assemblage de l’ADN », expliquent les chercheurs dans l’étude, publiée dans la revue Nature.
Un système inspiré des anciennes presses d’imprimerie
La technique consiste à encoder ces données binaires directement au niveau de l’ADN par le biais de la méthylation sélective – une modification épigénétique. Si la base est méthylée, elle est codée en 1, tandis qu’elle est codée en 0 si elle n’est pas méthylée. En d’autres termes, « ce n’est pas la séquence [ADN] elle-même qui porte l’information, mais plutôt la couche de méthylation », explique le coauteur de l’étude Long Qian, de l’Université de Pékin, au Chemical & Engineering News.
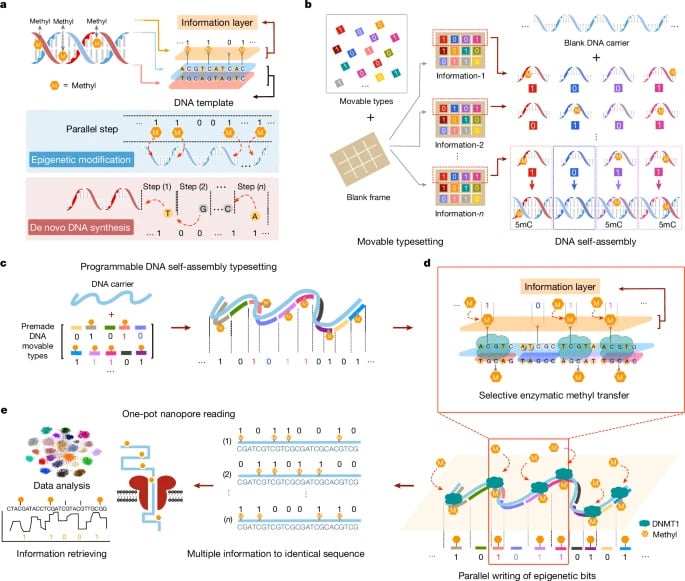
Les chercheurs ont développé un mode d’écriture programmable par auto-assemblage, similaire aux caractères mobiles utilisés dans les anciennes imprimeries. Pour imprimer les journaux, ces derniers étaient assemblés un par un sur de grandes plaques pour former les textes à imprimer, puis on y répandait de l’encre avant de passer le papier dessus. Pour la technique de l’étude, un modèle d’ADN monocaténaire (simple brin) universel fait office de papier, tandis que les caractères d’imprimerie sont un ensemble de 700 brins courts appelés « briques ». Chaque brique est composée de 24 nucléotides et les cytosines constituent les sites de méthylation.
Pour encoder les informations, les briques sont agencées au niveau du modèle d’ADN selon une séquence spécifique, chacune contenant un épibit d’information. L’ADN est traité avec de la méthyltransférase (une enzyme), qui reconnaît la cytosine méthylée et imprime les informations de méthylation en se liant au modèle d’ADN. Ce processus est comparable à la manière dont les données numériques sont copiées sur les disques durs en variant la direction de magnétisation. Les informations sont ensuite récupérées à l’aide d’une technique de séquençage par nanopores, permettant une lecture directe et en temps réel des fragments d’ADN et de leur état de méthylation.
L’équipe a testé sa technique d’impression sur ADN pour encoder un dessin représentant un tigre et la photo d’un panda, représentant respectivement 16 833 bits et 252 500 bits. L’ensemble des données a été imprimé et récupéré avec succès. Alors que les techniques précédentes nécessitaient entre 4 et 20 minutes pour ajouter les nucléotides un par un, cette avancée permet d’imprimer simultanément 350 bits d’information bien plus rapidement que les méthodes actuelles.
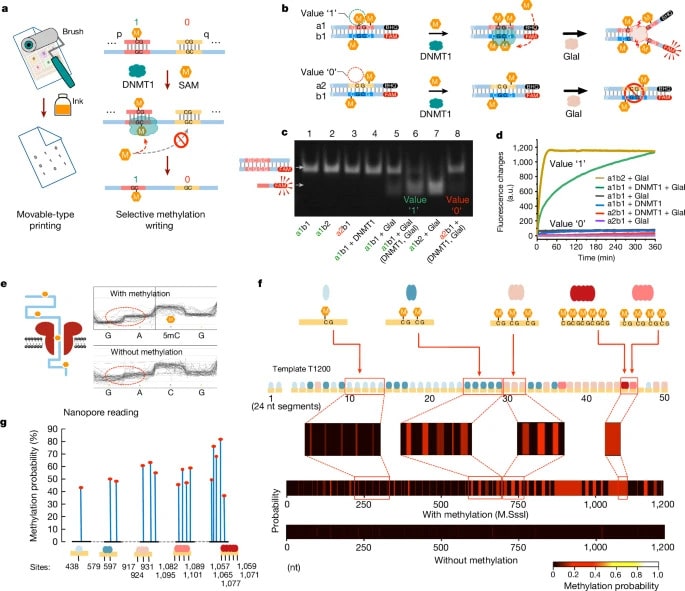


Accessible aux non-scientifiques et précise à 98,58 %
Afin d’évaluer l’accessibilité de la technique, les chercheurs ont invité 60 volontaires sans expérience en techniques de laboratoire à utiliser la plateforme de sauvegarde et de récupération de données. Les participants devaient utiliser un logiciel appelé iDNAdrive pour encoder des textes de leur choix et totalisant environ 5 000 bits. Alors que le stockage par nucléotide doit être effectué au niveau d’une plateforme de synthèse spécialisée et nécessite des compétences de laboratoire avancées, les manipulations nécessaires à cette nouvelle technique consistent simplement en des ajouts des divers réactifs dans un même tube. Les données ont été imprimées et lues avec une précision de 98,58 %.
Les chercheurs prévoient d’améliorer davantage la technique, notamment en se concentrant sur l’augmentation de la vitesse d’écriture des données et la réduction des coûts. Ils espèrent également augmenter la capacité de stockage en étendant le marquage épigénétique à d’autres bases nucléotidiques, plutôt que de se limiter uniquement à la cytosine. À terme, la technique pourrait potentiellement révolutionner le stockage de données, surtout en raison de son accessibilité.



