Si les modèles d’intelligence artificielle générative impressionnent par leur capacité à dialoguer avec les utilisateurs, leur potentiel dépasse largement le cadre de la conversation. Dans de nombreux domaines scientifiques, ces technologies offrent des solutions inédites, notamment dans le développement de nouvelles thérapies ou l’exploration des mécanismes biologiques. C’est précisément dans cette optique que des chercheurs ont développé un modèle multimodal, baptisé ESM3, qui a récemment été capable de générer une nouvelle protéine par un processus qui aurait nécessité près d’un demi-milliard d’années d’évolution naturelle. Une grande avancée qui pourrait mener à des applications intéressantes, notamment dans le développement de nouveaux médicaments.
« Si l’on pouvait remonter le cours de l’évolution et le rejouer, le résultat serait-il identique à celui que nous connaissons aujourd’hui, ou assisterions-nous à l’émergence d’un monde totalement différent ? ». Cette interrogation, formulée par le biologiste Stephen Jay Gould dans son ouvrage « Wonderful Life » (1989), alimente depuis des décennies les débats scientifiques.
Pour y répondre, certains chercheurs ont tenté de recréer l’évolution en laboratoire. C’est notamment le cas de l’expérience de Richard Lenski, qui observe depuis plus de trente ans les mutations et adaptations des bactéries Escherichia coli à travers des générations successives.
Aujourd’hui, l’essor de l’intelligence artificielle permet d’adopter une approche radicalement nouvelle. C’est ce qu’a entrepris la start-up américaine EvolutionaryScale, un laboratoire de recherche en IA spécialisé dans la biologie, soutenu par Amazon et Nvidia.
Une IA pour explorer l’évolution des protéines
Fondée il y a six ans et basée à New York, EvolutionaryScale s’est d’abord illustrée avec l’EvolutionaryScale Model 1 (ESM1), une IA générative multimodale conçue pour analyser et modéliser des protéines. Grâce à l’intégration massive de données biologiques – séquences et structures protéiques –, ESM1 a permis d’élucider la structure de centaines de millions de protéines issues du métagénome et s’est imposé comme un outil précieux pour la communauté scientifique.
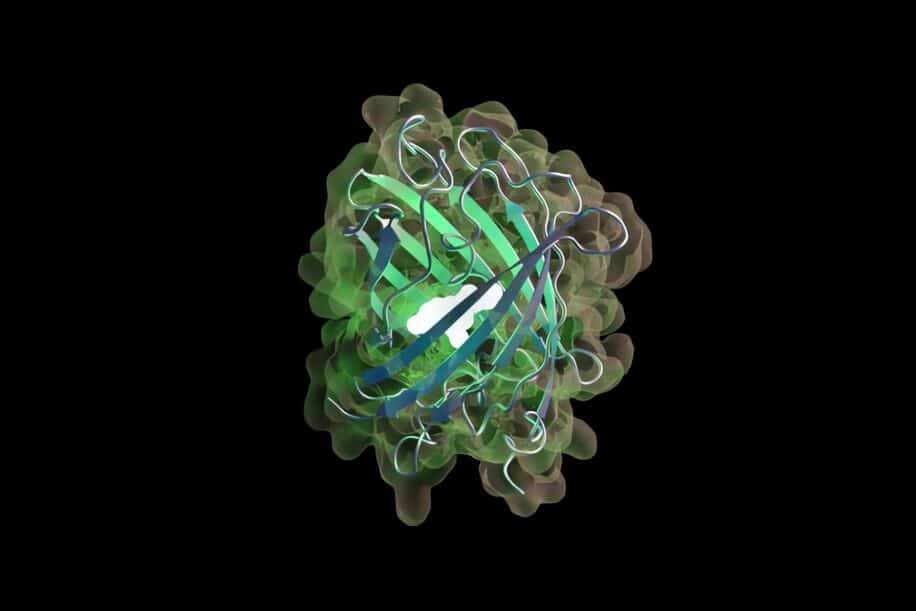
Forte de ce premier succès, l’équipe d’EvolutionaryScale a mis au point ESM3, une version plus avancée du modèle. À titre de démonstration, les chercheurs sont parvenus à générer une nouvelle protéine fluorescente verte, baptisée esmGFP, inspirée de la GFP (Green Fluorescent Protein) découverte chez les méduses. Les résultats préliminaires de cette recherche ont été présentés en juin 2024, avant d’être publiés ce mois-ci dans la revue Science.
Une puissance de calcul inédite au service de la biologie
Selon Alexander Rives, cofondateur et directeur scientifique d’EvolutionaryScale, ESM3 est le fruit de travaux initiés alors que lui et son équipe évoluaient encore au sein de Meta. « Ce modèle, comparable dans son principe à GPT-4, est spécifiquement entraîné pour comprendre la biologie », explique-t-il. L’ESM3 a été nourri d’une base colossale de 771 milliards de données, comprenant 3,15 milliards de séquences protéiques, 236 millions de structures et 539 millions de traits fonctionnels.
Avec une puissance de calcul de 1 000 milliards de téraflops, il surpasse largement tous les autres modèles existants dans le domaine de la biologie computationnelle. Son entraînement a nécessité l’exploitation de 2,78 milliards de protéines issues d’environnements variés, allant de la forêt amazonienne aux abysses océaniques, en passant par les sources hydrothermales et les micro-organismes du sol.
Pour évaluer ses performances, les chercheurs ont soumis ESM3 à un test particulier : il devait compléter des fragments manquants d’un schéma protéique en s’appuyant sur ses données d’apprentissage. Les résultats se sont révélés concluants. « Nous avons constaté qu’ESM3 intègre la biologie fondamentale et peut générer des protéines fonctionnelles situées au-delà des trajectoires explorées par l’évolution naturelle », confie Rives à Live Science. « Nos recherches montrent qu’en résolvant des tâches apparemment simples, des schémas biologiques profonds émergent spontanément dans le réseau », poursuit-il.
Dans leur étude, les chercheurs expliquent qu’ils ont spécifiquement entraîné ESM3 pour générer une nouvelle GFP. C’est ainsi que le modèle a produit esmGFP, qui présente 58 % de similarité avec sa plus proche cousine naturelle. Selon les chercheurs, cette avancée revient à simuler 500 millions d’années d’évolution.
Des applications prometteuses
Une telle découverte suggère que la nature aurait pu explorer d’autres trajectoires évolutives il y a un demi-milliard d’années, sans toutefois le faire. Les protéines fluorescentes auraient ainsi pu apparaître plus tôt, mais de subtiles mutations auraient pu inhiber cette évolution.
Avec de telles capacités, ESM3 pourrait être utilisé pour générer une vaste gamme de protéines aux fonctions thérapeutiques ou environnementales. Parmi les pistes envisagées figure la conception de protéines capables de dégrader les déchets plastiques ou d’accélérer certaines réactions biochimiques utiles. « Nous travaillons sur ce projet depuis longtemps et nous sommes impatients de voir comment la communauté scientifique s’en emparera », conclut Rives.