Un certain nombre de bactéries pathogènes à l’origine d’infections bactériennes engageant le pronostic vital chez l’humain utilisent le même mécanisme infectieux pour infiltrer les cellules et y délivrer des toxines. Une équipe de chercheurs est récemment parvenue à modéliser ce complexe dynamique infectieux avec un très haut niveau de détails. Les résultats devraient permettre de développer des antibiotiques ciblés plus précis et efficaces.
Cancer gastrique, fièvre Q, légionellose, coqueluche — bien que les bactéries pathogènes à l’origine de ces maladies dangereuses soient différentes, elles utilisent toutes le même mécanisme moléculaire pour infecter les cellules humaines. Les bactéries utilisent ce mécanisme, appelé système de sécrétion de type IV (T4SS), pour injecter des molécules toxiques dans les cellules, et également pour diffuser des gènes de résistance aux antibiotiques à d’autres bactéries.
Des chercheurs du Caltech ont révélé l’architecture moléculaire 3D du T4SS issu du pathogène humain Legionella pneumophila, avec des détails sans précédent. Cela pourrait à l’avenir permettre le développement d’antibiotiques ciblés avec précision pour les maladies susmentionnées. La recherche a été publiée dans la revue Nature Microbiology.
Une modélisation ultra-détaillée du complexe d’infection bactérien
Il existe neuf types différents de systèmes de sécrétion bactérienne, le type IV étant le plus élaboré et le plus polyvalent. Un T4SS peut transporter une grande variété de molécules toxiques — jusqu’à 300 en même temps — d’une bactérie à une cible cellulaire, en détournant des fonctions cellulaires et en surchargeant les défenses de la cellule.
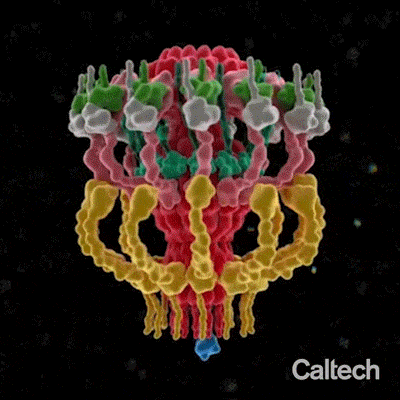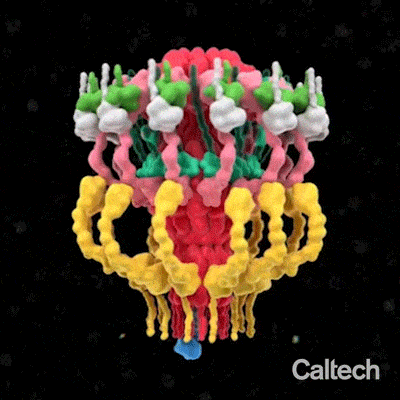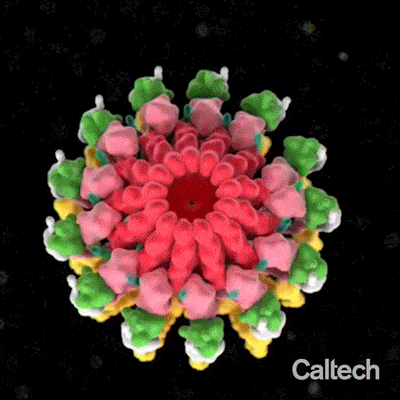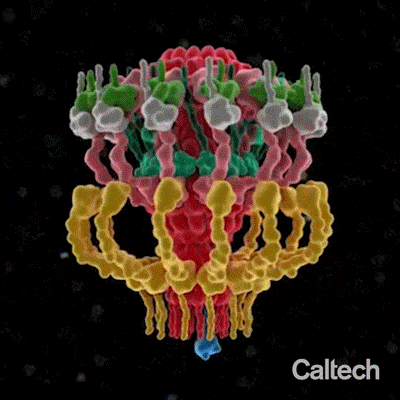
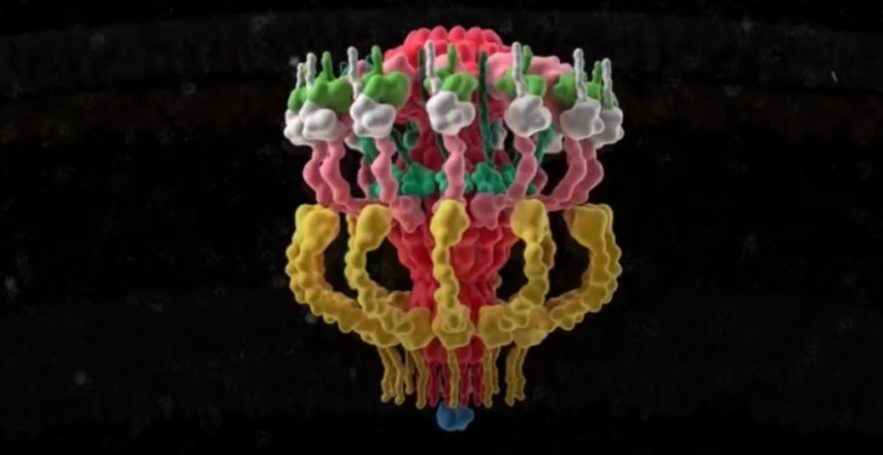
En 2017, Debnath Ghosal, chercheur au Caltech, et ses collaborateurs ont utilisé une technique appelée cryotomographie électronique pour révéler, pour la première fois, l’architecture globale à basse résolution du T4SS dans Legionella, la bactérie qui cause la maladie du légionnaire, une maladie grave et souvent mortelle.
Sur le même sujet : Découverte d’un nouveau gène bactérien de résistance à un antibiotique de dernier recours
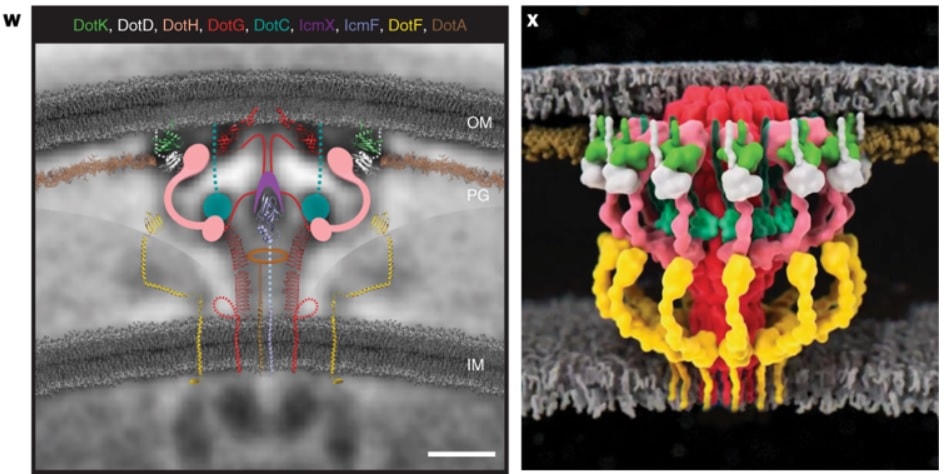
Ghosal, avec Kwangcheol Jeong du WUSTL et leurs collègues, ont maintenant élaboré un modèle structurel détaillé de cette machine dynamique à plusieurs composants. L’équipe a également procédé à des perturbations précises des gènes de la bactérie afin d’étudier les versions mutantes du T4SS, révélant comment cette machine complexe s’organise et s’assemble.
Des pistes pour le développement d’antibiotiques plus précis et efficaces
Le modèle a révélé que le système de sécrétion est composé d’une chambre distincte et d’un long canal, comme la chambre et le canon d’un fusil. La caractérisation de ces composants ainsi que d’autres composants du T4SS pourrait permettre le développement d’antibiotiques ciblés avec précision.
Les antibiotiques actuels agissent largement et éliminent les bactéries dans tout le corps, y compris les micro-organismes bénéfiques qui vivent dans nos intestins. À l’avenir, les antibiotiques pourraient être conçus pour bloquer uniquement les systèmes d’administration de toxines (tels que le T4SS) d’agents pathogènes nocifs, rendant les bactéries inertes et inoffensives sans perturber les bonnes bactéries.