Au cours d’une vie, l’ADN subit naturellement des dommages mineurs constants, nécessitant une réparation rapide et proche de la perfection. La réparation de ces cassures d’ADN est critique pour un organisme, car même les modifications les plus simples d’une séquence risquent d’être catastrophiques, surtout si le code altéré est responsable d’une fonction importante. Récemment, des chercheurs ont observé en détail et en temps réel des bactéries en train de réparer de l’ADN endommagé.
Depuis des décennies, les biologistes étudient les mécanismes impliqués pour reconstituer la plupart des grandes étapes de la réparation de l’ADN. Pourtant, une partie du processus est restée inexplorée, faute de technologies adaptées à son étude détaillée en temps réel.
Récemment, en marquant les enzymes clés et l’ADN avec des marqueurs fluorescents et en observant le processus de réparation en temps réel dans un modèle d’Escherichia coli, des chercheurs de l’université d’Uppsala, en Suède, ont complété les détails manquants sur la façon dont les bactéries trouvent les modèles sur lesquels elles comptent pour que les réparations génétiques soient exemptes d’erreurs. L’étude a été publiée dans la revue Nature.
Processus de recombinaison homologue : préserver l’ADN
L’une des astuces utilisées par la plupart des êtres vivants pour maintenir leur code génétique intact est le processus de recombinaison homologue, l’équivalent biologique de la comparaison de deux versions distinctes d’un script pour s’assurer qu’une copie n’a pas introduit d’erreur. En conservant une version non endommagée d’une séquence durant un travail de réparation, une cellule peut s’assurer qu’aucun changement n’a eu lieu lorsque les extrémités coupées sont collées ensemble.
Les biologistes moléculaires savent depuis longtemps que la protéine recombinase RecA joue un rôle clé dans la gestion de ce processus. C’est une enzyme si importante pour le maintien de l’intégrité de l’ADN qu’on en a trouvé une version dans pratiquement toutes les espèces étudiées.
Lorsqu’une « échelle » d’ADN double brin se rompt complètement, un complexe de protéines se met au travail pour saisir les extrémités coupées et les tailler proprement afin que RecA puisse s’installer et faire son travail. La protéine s’étend alors en un long faisceau, formant un filament de protéine et d’acide nucléique capable de s’accrocher à la fois au brin cassé et à une deuxième échelle intacte d’ADN non cassé.
À partir de là, le filament doit trouver la bonne séquence pour servir de point de comparaison. La manière dont le filament parvient à effectuer cette recherche en un temps suffisamment court est un mystère depuis près de 50 ans, d’autant plus que des millions de paires de bases doivent être vérifiées dans les méandres complexes du chromosome.
Une réparation homologique rapide et réussie grâce à RecA
Pour mieux comprendre le timing et la navigation de l’enzyme à l’œuvre, les chercheurs ont cultivé des milliers de cellules d’E. coli à l’intérieur d’une série de minuscules canaux qui leur ont permis de suivre les bactéries individuelles au fur et à mesure de leurs expériences.
Une fois les cellules en place, les scientifiques ont effectué des cassures précises dans leur ADN à l’aide de la technique d’édition de gènes CRISPR, marquant les extrémités coupées avec des marqueurs fluorescents pour visualiser l’emplacement de la cassure au microscope. « La puce de culture microfluidique nous permet de suivre l’évolution de milliers de bactéries individuelles simultanément et de contrôler les cassures d’ADN induites par CRISPR à temps », explique Jakub Wiktor, biologiste moléculaire de l’université d’Uppsala.
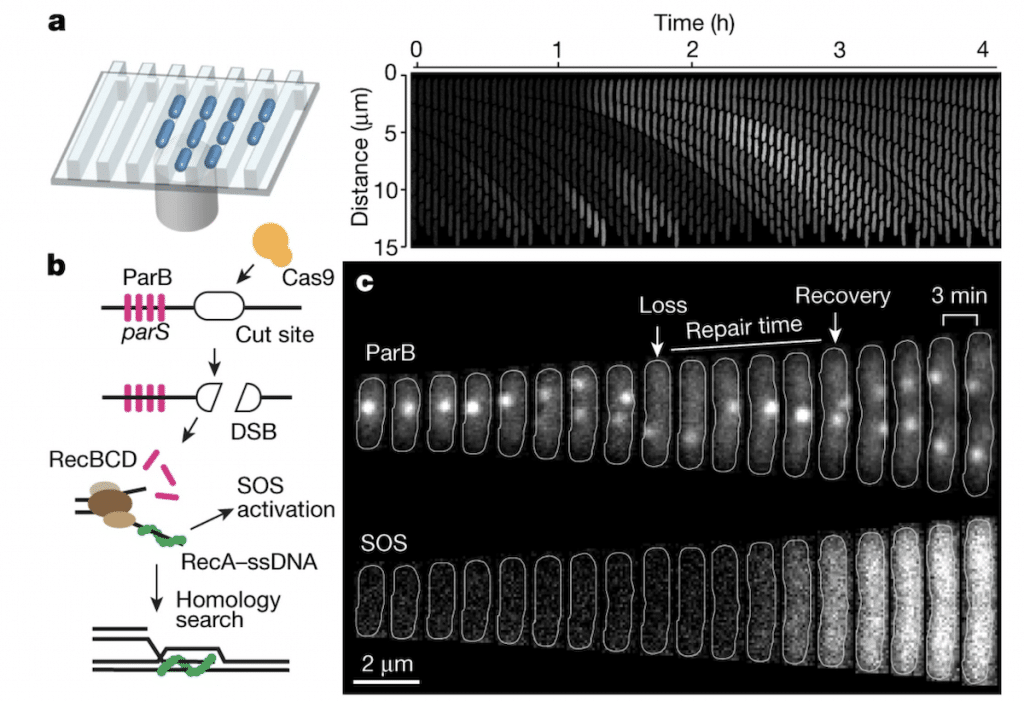
Enfin, ils ont utilisé des anticorps pour identifier l’emplacement des filaments de RecA au fur et à mesure qu’ils se mettent en place et poursuivent leur recherche, comme dans une bibliothèque. Une indicateur chimique a indiqué à l’équipe quand l’ensemble du processus de réparation était terminé. En moyenne, il n’a fallu que 15 minutes à E. coli pour terminer son travail. Étonnamment, il ne fallait que 9 minutes pour que la protéine trouve le bon modèle.
Le secret semble résider dans la construction du filament nucléoprotéique de RecA. Ce fil s’étend à travers la cellule, s’accroche au chromosome et glisse vers le bas à la recherche d’une correspondance avec la séquence qu’il saisit. Bien que cela ne semble pas très efficace, ce n’est pas vraiment différent que de parcourir méthodiquement les allées d’une bibliothèque à la recherche d’un livre correspondant à la cote du catalogue.
« Puisque les extrémités de l’ADN sont incorporées dans cette fibre, il suffit que n’importe quelle partie du filament trouve le précieux gabarit, et la recherche est donc théoriquement réduite de trois à deux dimensions », explique Arvid Gynnå. « Notre modèle suggère que c’est la clé d’une réparation homologique rapide et réussie ».
Bien que cette étude ait été menée sur des bactéries, le fait que RecA soit si similaire dans toute la biosphère permet déjà de prédire son fonctionnement dans le corps humain. Maintenant que nous en savons plus sur le processus complet, nous pouvons commencer à rechercher les signes de situations où la réparation de notre propre ADN se déroule incorrectement, ouvrant ainsi la voie à la compréhension des origines de maladies graves comme le cancer.