À la recherche d’agents pharmaceutiques, comme de nouveaux vaccins, l’industrie pharmaceutique analyse régulièrement des milliers de molécules candidates et apparentées. Accélérer le processus est une nécessité en temps de pandémie, comme nous en avons fait l’expérience avec la COVID-19. Récemment, une équipe de chercheurs danois a mis au point une nouvelle technique de synthèse et de criblage de molécules à l’échelle nanométrique, en minimisant l’utilisation de matériaux et d’énergie. Plus de 40 000 molécules différentes peuvent être synthétisées et analysées dans des nanoconteneurs plus petits qu’une tête d’épingle (similaires à des bulles de savon) en seulement 7 min.
Avec le séquençage systématique du génome humain et d’agents pathogènes, le nombre de cibles potentielles de médicaments a augmenté de manière exponentielle au cours des années 1990. Paradoxalement, les informations disponibles pour chacune de ces nouvelles cibles — récepteurs, enzymes ou éléments de régulation de l’expression des gènes — sont peu nombreuses. Dans certains cas, elles peuvent se réduire à la simple séquence nucléotidique d’un gène. Face à cette situation, l’industrie pharmaceutique utilise la méthode de criblage systématique « en aveugle » de grandes collections de composés (chimiothèques).
Ce criblage à haut débit consiste à faire réagir simultanément un grand nombre de molécules différentes avec un substrat donné, en vue d’identifier, en un minimum de temps, celles de ces molécules qui présentent un intérêt éventuel pour une application déterminée. L’utilisation massive de la robotique et de l’informatique, jointe à la miniaturisation des tests biochimiques, a permis d’augmenter considérablement le nombre de composés susceptibles d’être testés quotidiennement.
Cependant, les chimiothèques historiques ne contiennent pas suffisamment de molécules à tester, surtout lorsque les chercheurs ne connaissent pas précisément ce qu’ils doivent trouver, par manque d’informations sur les cibles. C’est pourquoi les chimistes ont mis au point de nouvelles stratégies permettant de synthétiser, à la demande, de grandes séries de composés de structures différentes, adaptées aux exigences du criblage systématique.
Cette chimie combinatoire s’adapte aux programmes de recherche en partant de la constatation que : moins nous avons d’informations sur la cible à cribler, plus la diversité requise pour la chimiothèque et le nombre de molécules à synthétiser est grande. Ces méthodologies combinatoires à haut débit sont essentielles à la fois pour le criblage et la découverte en biochimie synthétique et en sciences biomédicales. Néanmoins, ils dépendent souvent d’analyses à grande échelle et sont donc limités par une longue durée de fonctionnement et un coût excessif des matériaux.
C’est dans ce contexte que récemment, une équipe interdisciplinaire de l’Université de Copenhague au Danemark a développé une nouvelle technique de criblage à haut débit, en se basant sur la nanotechnologie de l’ADN, accélérant le processus d’un million de fois, réduisant de fait les coûts associés. Leurs travaux sont publiés dans Nature Chemistry.
Des bulles de savon pour un criblage à haut débit en 7 min
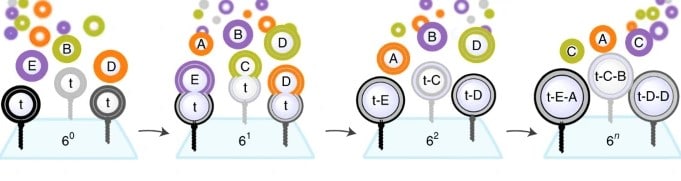
Le travail a été réalisé en collaboration entre le groupe Hatzakis de l’Université de Copenhague et le professeur associé Stefan Vogel de l’Université du Danemark du Sud. La technique, surnommée « fusion de nanoconteneurs lipidiques combinatoires à une seule particule basée sur la fusion médiée par l’ADN » ou SPARCLD, utilise de minuscules « bulles » ressemblant à du savon. Ces bulles forment des « nano-conteneurs » à l’intérieur desquels des molécules peuvent être produites grâce à la nanotechnologie de l’ADN. Environ 42 000 nanoconteneurs peuvent tenir sur un millimètre carré.
Certes, comme le précise, dans un communiqué, Nikos Hatzakis, chef d’équipe et professeur associé au département de chimie de l’université de Copenhague : « Aucun élément de notre solution n’est complètement nouveau, mais ils n’ont jamais été combinés de manière aussi transparente ». Plus précisément, la technologie intègre des éléments issus de disciplines normalement assez éloignées : la biochimie de synthèse, les nanotechnologies, la synthèse d’ADN, la chimie combinatoire, et même le Machine Learning — discipline de l’intelligence artificielle.

C’est ainsi que certaines des « bulles », ou liposomes, sont attachées à une surface, tandis que d’autres flottent librement. Chaque bulle contient une séquence d’ADN différente et des marqueurs fluorescents pouvant être détectés par un microscope à réflexion interne totale en temps réel (TIRF). Au fur et à mesure que les bulles flottent et fusionnent de manière aléatoire, de nombreuses combinaisons différentes de fragments d’ADN peuvent être créées et détectées en temps réel à l’aide du microscope.
D’ailleurs, les chercheurs ont observé des séquences de fusion efficaces à plus de 93%. Par suite, un algorithme d’apprentissage automatique décode les images de microscopie pour classer les « séquences de fusion » créées. Ainsi, la microscopie en temps réel a permis l’observation directe de plus de 16 000 fusions et une classification précise de 566 séquences de fusion distinctes. Les résultats sont connus en seulement 7 min.
La doctorante Mette G. Malle, auteure principale de l’article et actuellement chercheuse postdoctorale à l’Université de Harvard, aux États-Unis, déclare : « Ce que nous avons est très proche d’une lecture en direct. Cela signifie que l’on peut modérer la configuration en continu en fonction des lectures en ajoutant une valeur supplémentaire significative. Nous nous attendons à ce que ce soit un facteur clé pour l’industrie qui souhaite mettre en œuvre la solution ».
Développement de produits pharmaceutiques à coûts réduits
Les chercheurs sont enthousiasmés par le potentiel de la technologie permettant un criblage extrêmement rapide et efficace, de milliers de molécules, candidates pour des applications telles que la production de vaccins et de produits pharmaceutiques. Nikos Hatzakis souligne : « Il s’agit d’une économie d’efforts, de matériel, de main-d’œuvre et d’énergie sans précédent ».
Malgré cet enthousiasme, l’équipe a dû garder sa découverte sous silence, jusqu’à la parution de l’article. En effet, les chercheurs du projet ont, individuellement, plusieurs collaborations industrielles. Les risques de fuites et de plagiat étaient alors trop grands. Néanmoins, ils ne savent toujours pas quelles entreprises pourraient souhaiter mettre en œuvre la nouvelle méthode à haut débit.
Quoi qu’il en soit, à l’heure actuelle, il s’agit de diffuser cette technique SPARCLD à travers de nombreuses applications potentielles dans la recherche et l’industrie. Par exemple, elle pourrait être utilisée pour synthétiser et cribler des molécules d’ARN à utiliser dans l’édition de gènes médiée par CRISPR. Ce dernier donne la possibilité de couper des séquences d’ADN impliquées dans une mutation et les remplacer par des séquences d’ADN corrigeant la mutation. SPARCLD pourrait également être utilisé pour le développement de futurs vaccins à ARN.
Nikos Hatzakis déclare : « Il y a fort à parier que les groupes industriels et universitaires impliqués dans la synthèse de longues molécules telles que les polymères pourraient être parmi les premiers à adopter la méthode ». Il conclut « Notre configuration permet d’intégrer SPARCLD avec une lecture post-combinatoire pour des combinaisons de réactions protéine-ligand telles que celles pertinentes pour une utilisation dans CRISPR. Seulement, nous n’avons pas encore pu aborder cela, puisque nous voulions d’abord publier notre méthodologie ».