Malgré l’essor des outils d’ingénierie du génome au cours de la dernière décennie, les chercheurs manquent toujours de méthodes fiables pour l’insertion précise de grandes séquences d’ADN. S’appuyant sur le système d’édition de gènes CRISPR, les chercheurs du MIT ont conçu un nouvel outil capable de supprimer de grands gènes défectueux et de les remplacer par de nouveaux, de manière plus sûre et plus efficace. La technique a ainsi le potentiel de traiter une variété de maladies génétiques.
La première génération de CRISPR pouvait couper l’ADN, la deuxième génération permettait l’édition de base, et avec la troisième génération est venue l’édition Prime — la possibilité d’apporter de petites modifications (insertions et suppressions de paires de bases).
Mais les chercheurs étaient encore limités à de petites délétions et insertions, laissant un vide de plusieurs kilobases dans le domaine de l’édition de gènes. Le développement, par une équipe du MIT, d’une quatrième technique pallie ces défauts. Nommée PASTE, celle-ci peut « glisser-déposer » la valeur de kilobases d’ADN avec une précision basée sur CRISPR, dans un génome.
Récemment, en utilisant cette méthode, ces mêmes chercheurs ont montré qu’ils pouvaient délivrer des gènes aussi longs que 36 000 paires de bases d’ADN à plusieurs types de cellules humaines, ainsi qu’aux cellules hépatiques de souris, sans abîmer l’ADN. Cette nouvelle technique pourrait être prometteuse pour le traitement de maladies causées par des gènes défectueux avec un grand nombre de mutations, comme la fibrose kystique. Leur étude est publiée dans la revue Nature Biotechnology.
Glisser-déposer au lieu de couper-coller
Il faut savoir que le système d’édition de gènes CRISPR-Cas9 se compose d’une enzyme coupant l’ADN appelée Cas9 et d’un court brin d’ARN qui guide l’enzyme vers la zone spécifique du génome à couper. Lorsque Cas9 et l’ARN guide ciblant un gène de la maladie sont délivrés dans les cellules, une coupe spécifique est effectuée dans le génome et les processus de réparation de l’ADN des cellules collent les deux extrémités ensemble, supprimant souvent une petite partie du génome.
Afin d’éviter cette perte d’information, les chercheurs peuvent délivrer, en même temps que la coupe, une séquence d’ADN à incorporer (comme un couper-coller). Mais cette technique abîme l’ADN ciblé en provoquant des cassures double brin, ce qui peut provoquer des délétions ou des réarrangements chromosomiques nocifs pour les cellules.
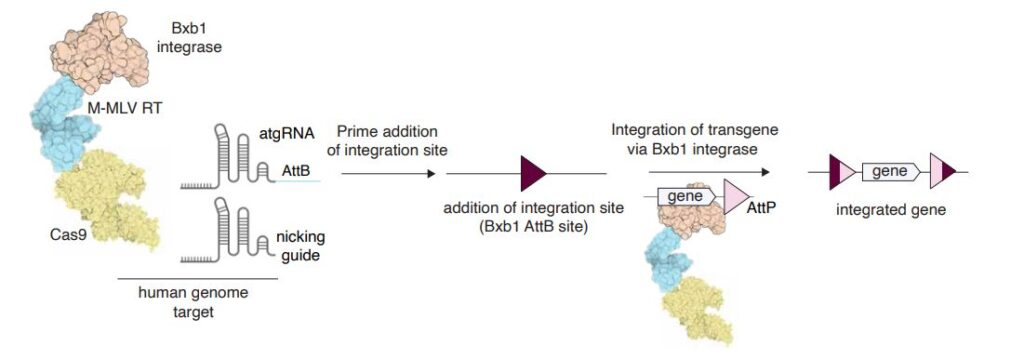
Par conséquent, le nouvel outil combine le ciblage précis de CRISPR-Cas9 avec des enzymes appelées intégrases, que les virus utilisent pour insérer leur propre matériel génétique dans un génome bactérien.
Pour cette étude, les chercheurs se sont concentrés sur les intégrases à sérine, qui peuvent insérer d’énormes morceaux d’ADN, pouvant atteindre 50 000 paires de bases. Ces enzymes ciblent des séquences génomiques spécifiques appelées sites de fixation, qui fonctionnent comme des « patins d’atterrissage », selon les auteurs. Lorsqu’ils trouvent la bonne plateforme d’atterrissage dans le génome de l’hôte, ils s’y lient et intègrent leur charge utile d’ADN.
Le nouvel outil, PASTE (Programmable Addition via Site-specific Targeting Elements), comprend donc une enzyme Cas9 qui coupe sur un site génomique spécifique, guidée par un brin d’ARN qui se lie à ce site. Cela leur permet de cibler n’importe quel endroit du génome pour l’insertion du site d’atterrissage, qui contient 46 paires de bases d’ADN. Comme il s’agit d’un petit fragment, l’insertion ne provoque aucune cassure double brin.
Une fois le site d’atterrissage incorporé, l’intégrase peut arriver et insérer sa charge utile d’ADN beaucoup plus grande dans le génome de ce site.
Jonathan Gootenberg, membre du McGovern Institute for Brain Research du MIT, déclare dans un communiqué : « Nous pensons qu’il s’agit d’un grand pas vers la réalisation du rêve d’une insertion programmable d’ADN. C’est une technique qui peut être facilement adaptée à la fois au site que nous voulons intégrer et à la cargaison ».
Espoir partagé pour de nombreuses maladies
Dans cette étude, les chercheurs ont montré qu’ils pouvaient utiliser PASTE pour insérer des gènes dans plusieurs types de cellules humaines, notamment les cellules hépatiques, les cellules T et les lymphoblastes (globules blancs immatures).
Ils ont testé le système de délivrance avec 13 gènes de charge utile différents, dont certains pourraient être utiles sur le plan thérapeutique, et ont pu les insérer à neuf endroits différents du génome. Dans ces cellules, les chercheurs ont pu insérer des gènes avec un taux de réussite allant de 5 à 60%.
Les chercheurs ont également démontré qu’ils pouvaient insérer des gènes dans des foies « humanisés » de souris, constitués d’environ 70% d’hépatocytes humains. PASTE a réussi à intégrer de nouveaux gènes dans environ 2,5% de ces cellules.
Actuellement, les chercheurs explorent davantage la possibilité d’utiliser cet outil comme moyen de remplacer le gène défectueux de la fibrose kystique. Cette technique pourrait également être utile pour traiter les maladies du sang causées par des gènes défectueux, comme l’hémophilie et le déficit en G6PD, ou la maladie de Huntington.
Les chercheurs ont rendu leurs constructions génétiques disponibles en ligne pour que d’autres scientifiques puissent les utiliser. En effet, les séquences d’ADN que les chercheurs ont insérées dans cette étude comportaient jusqu’à 36 000 paires de bases, mais ils pensent que des séquences encore plus longues pourraient également être utilisées.
Gootenberg conclut : « L’un des aspects fantastiques de l’ingénierie de ces technologies moléculaires est que les gens peuvent s’appuyer sur elles, les développer et les appliquer d’une manière à laquelle nous n’avions peut-être pas pensé. C’est vraiment formidable de faire partie de cette communauté émergente ».



