Depuis des mois, des nouvelles variantes plus contagieuses du coronavirus SARS-CoV-2 — en provenance notamment du Royaume-Uni, de l’Afrique du Sud et du Brésil, se propagent rapidement à travers le monde, et les chercheurs tentent depuis de comprendre ce qui rend ces dernières si infectieuses. Une série d’études menées à l’hôpital pour enfants de Boston a analysé comment la structure des protéines S (Spike) du coronavirus affecte la capacité d’infection de la mutation D614G (présente dans les trois variantes), et les résultats ont révélé pourquoi ces variantes sont capables de se propager plus rapidement.
L’équipe, dirigée par Bing Chen, a imagé les protéines S par cryo-microscopie électronique (cryo-EM), avec une résolution allant jusqu’au niveau atomique. Ils ont alors découvert que la mutation D614G (substitution d’une seule « lettre » d’acide aminé dans le code génétique de la protéine S) rend l’ensemble de la protéine plus stable par rapport au SARS-CoV-2 original. Par conséquent, un plus grand nombre de protéines S fonctionnelles sont disponibles pour se lier aux récepteurs ACE2 de nos cellules, rendant le virus plus infectieux. Les détails ont été publiés dans la revue Science le 16 mars 2020.
Une protéine S plus stable
Dans la souche originale du SARS-CoV-2, les protéines S se liaient au récepteur ACE2 puis changeaient radicalement de forme en se repliant sur elles-mêmes. Cela permettait au virus de fusionner sa membrane avec celle de nos propres cellules et d’y pénétrer. Cependant, comme Chen et ses collègues l’ont rapporté dans une précédente étude, les protéines S changeaient parfois prématurément de forme et se désagrégeaient avant que le virus ne puisse se lier aux cellules. Si cela ralentissait la réplication du virus, le changement de forme rendait également plus difficile pour notre système immunitaire de contenir le virus. « Comme la protéine de pointe originale se dissociait, elle n’était pas assez fonctionnelle pour induire une forte réponse d’anticorps neutralisants », explique Chen.
Lorsque Chen et ses collègues ont imagé la protéine S mutante, ils ont découvert que la mutation D614G la stabilise en bloquant le changement de forme prématuré. La mutation fait également que la protéine S se lie plus faiblement au récepteur ACE, mais le fait qu’elle soit moins susceptible de se désagréger prématurément rend tout de même le virus globalement plus infectieux.
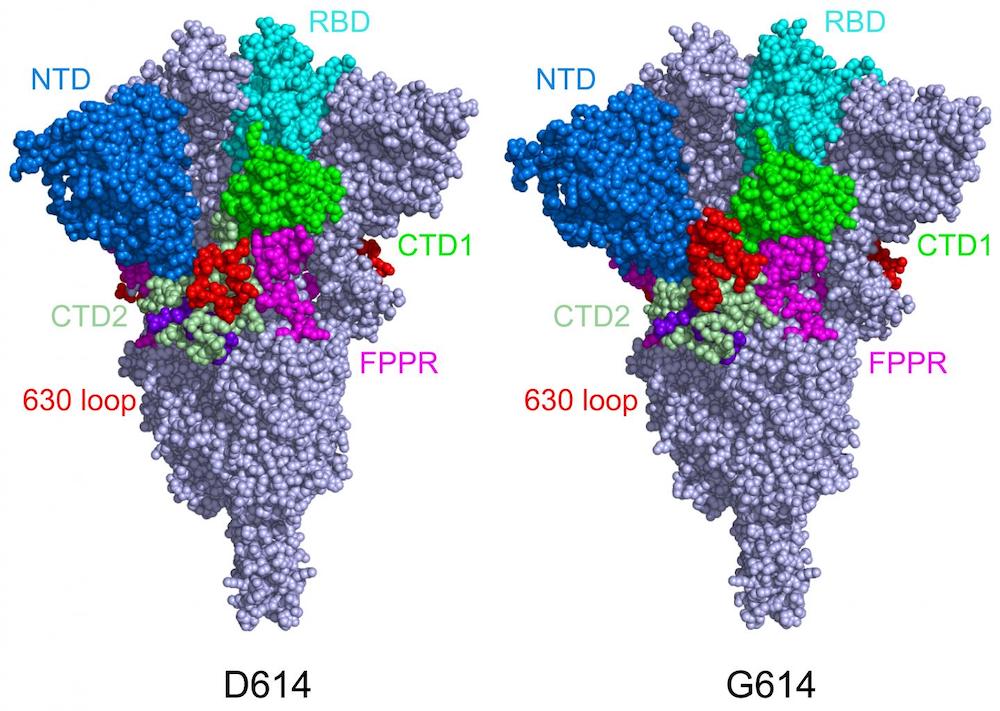
« Supposons que le virus original comporte 100 protéines S », explique Chen. « En raison de l’instabilité de la forme, seuls 50% d’entre elles peuvent être fonctionnelles. Dans les variants G614, 90% sont fonctionnelles et, même si elles ne se lient pas aussi bien, les chances d’être infecté sont plus grandes ».
Et concernant l’efficacité des vaccins ? Chen propose que les vaccins remaniés intègrent le code génétique de cette protéine S mutante. Cette forme plus stable devrait rendre tout vaccin basé sur la protéine S (comme les vaccins Moderna, Pfizer et Johnson & Johnson) plus susceptible de susciter des anticorps neutralisants protecteurs, explique-t-il. « Ces résultats élargissent notre compréhension de l’entrée virale et suggèrent un immunogène amélioré pour le développement de vaccins », écrivent les chercheurs dans le document.
Développement d’un médicament pour bloquer l’infection
Actuellement, Chen et ses collègues poursuivent l’application de la biologie structurelle pour mieux comprendre comment le SARS-CoV-2 se lie au récepteur ACE2 des cellules, dans l’optique de développer un traitement visant à empêcher le virus d’y pénétrer.
En janvier, l’équipe de Chen a publié un article dans la revue Nature Structural & Molecular Biology montrant qu’une protéine ACE2 « leurre » qu’ils ont conçue est capable de se lier au virus 200 fois plus fortement que l’ACE2 de l’organisme. Ainsi, le leurre a puissamment inhibé le virus en culture cellulaire, ce qui laisse penser qu’il pourrait constituer un traitement anti-COVID-19 efficace. Chen prévoit maintenant d’étendre cette recherche à des modèles animaux.