En décembre 2020, DeepMind a surpris le monde de la biologie lorsqu’il a résolu un grand défi de 50 ans avec AlphaFold, une IA qui prédit la structure des protéines. La semaine dernière, la société basée à Londres a publié tous les détails de cet outil et publié son code source. La société a récemment annoncé qu’elle avait utilisé son IA pour prédire les formes de presque toutes les protéines du corps humain, ainsi que les formes de centaines de milliers d’autres protéines trouvées dans 20 des organismes les plus étudiés, y compris la levure, les drosophiles et les souris.
Cette percée pourrait permettre aux biologistes du monde entier de mieux comprendre les maladies et de développer de nouveaux médicaments. Jusqu’à présent, la base de données se compose de 350 000 structures protéiques nouvellement prédites. DeepMind doit prédire et publier les structures de plus de 100 millions de protéines supplémentaires au cours des prochains mois, soit plus ou moins toutes les protéines connues de la science.
« Le repliement des protéines est un problème que je surveille depuis plus de 20 ans. Cela a été un énorme projet pour nous. Je dirais que c’est notre plus grand accomplissement jusqu’à présent. Et c’est le plus excitant d’une certaine manière, car il devrait avoir le plus grand impact dans le monde en dehors de l’IA », déclare Demis Hassabis, cofondateur et PDG de DeepMind.
Un outil de prédiction essentiel à la recherche
Les protéines sont constituées de longs rubans d’acides aminés, qui se tordent en nœuds compliqués. Connaître la forme du nœud d’une protéine peut révéler ce que fait cette protéine, ce qui est crucial pour comprendre le fonctionnement des maladies et développer de nouveaux médicaments, ou identifier des organismes qui peuvent aider à lutter contre la pollution et le changement climatique. Déterminer la forme d’une protéine prend des semaines ou des mois en laboratoire. AlphaFold peut prédire des formes à l’atome le plus proche en un jour ou deux.
La nouvelle base de données devrait faciliter encore plus la vie des biologistes. AlphaFold peut être disponible pour les chercheurs, mais tout le monde ne pourra pas exécuter le logiciel lui-même. « Il est beaucoup plus facile de récupérer une structure dans la base de données que de l’exécuter sur votre propre ordinateur », explique David Baker de l’Institute for Protein Design de l’Université de Washington, dont le laboratoire a développé son propre outil pour prédire la structure des protéines, appelé RoseTTAFold, basé sur l’approche d’AlphaFold.
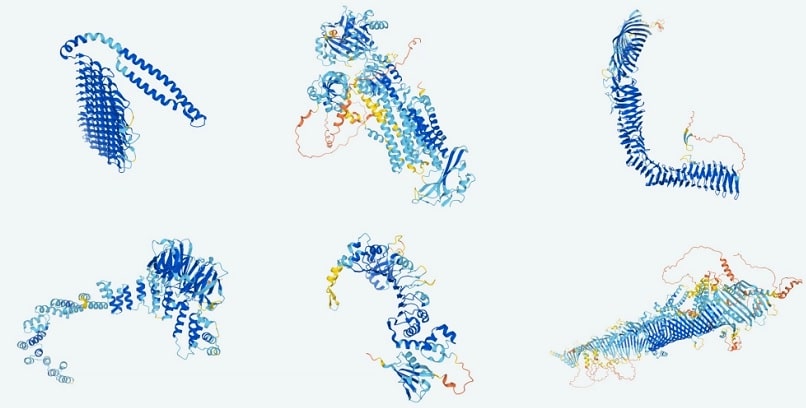
Au cours des derniers mois, l’équipe de Baker a travaillé avec des biologistes qui étaient auparavant coincés à essayer de comprendre la forme des protéines qu’ils étudiaient. « Il y a beaucoup de recherches biologiques assez intéressantes qui ont été vraiment accélérées », déclare Baker. Une base de données publique contenant des centaines de milliers de formes de protéines prêtes à l’emploi devrait être un accélérateur encore plus important. Cela semble étonnamment impressionnant, selon Tom Ellis, biologiste synthétique à l’Imperial College de Londres. Mais il prévient que la plupart des formes prédites n’ont pas encore été vérifiées en laboratoire.
Une précision à l’échelle atomique
Dans la nouvelle version d’AlphaFold, les prédictions sont accompagnées d’un score de confiance que l’outil utilise pour indiquer à quel point il estime que chaque forme prédite est proche de la réalité. À l’aide de cette mesure, DeepMind a découvert qu’AlphaFold prédisait les formes de 36 % des protéines humaines avec une précision correcte jusqu’au niveau des atomes individuels. C’est suffisamment précis pour le développement de médicaments, indique Hassabis. Auparavant, après des décennies de travail, seulement 17 % des protéines du corps humain avaient vu leurs structures identifiées en laboratoire.
Si les prédictions d’AlphaFold sont aussi précises que le prétend DeepMind, l’outil aurait plus que doublé ce nombre en quelques semaines seulement. Même les prédictions qui ne sont pas entièrement précises au niveau atomique sont toujours utiles. Pour plus de la moitié des protéines du corps humain, AlphaFold a prédit une forme qui devrait être suffisamment bonne pour que les chercheurs puissent comprendre la fonction de la protéine. Le reste des prédictions actuelles d’AlphaFold sont soit incorrectes, soit concernent le tiers des protéines du corps humain qui n’ont aucune structure jusqu’à ce qu’elles se lient aux autres.
« Le fait qu’il puisse être appliqué à ce niveau de qualité est impressionnant », déclare Mohammed AlQuraish, biologiste des systèmes à l’Université Columbia, qui a développé son propre logiciel pour prédire la structure des protéines. Il souligne également que le fait d’avoir des structures pour la plupart des protéines d’un organisme permettra d’étudier comment ces protéines fonctionnent en tant que système, et pas seulement de manière isolée. « C’est ce qui me semble le plus excitant ».