Des chercheurs ont mis au point une nouvelle technique d’édition génétique permettant de remodeler complètement le génome. Appelée « édition pont », elle permet entre autres d’induire des changements bien plus importants que ce qui est actuellement possible avec la technique CRISPR, notamment en insérant directement de nouvelles séquences d’ADN au sein du génome. Cela lui confère un niveau de précision jamais obtenu avec les techniques d’édition actuelles.
Les systèmes guidés par ARN ont révolutionné notre compréhension du génome. Parmi ces systèmes figure la technique d’édition génétique CRISPR, qui fonctionne comme des « ciseaux » moléculaires guidés par des ARN guides (ARNg). La technique permet entre autres de sectionner et de modifier une séquence génétique spécifique en personnalisant l’ARNg.
L’ensemble du processus est induit par la protéine CRISPR Cas. Le terme Cas désigne une nucléase qui se lie à un court ARN CRISPR (ARNcr) ciblant les séquences d’ADN ou d’ARN concernées. Les endonucléases Cas9 et Cas12 clivent l’ADN, tandis que Cas13 clive l’ARN. Lorsque l’outil moléculaire clive la séquence à cibler, la cellule induit systématiquement un processus de réparation. Le clivage est effectué à plusieurs reprises jusqu’à ce que la cellule provoque naturellement une mutation en induisant une erreur de réparation.
Cependant, bien que la technique CRISPR permette d’induire une mutation au niveau de sites spécifiques, son niveau de précision est relativement limité. Elle coupe et endommage les séquences cibles pour les inactiver, ce qui en fait davantage une technique destructrice qu’éditrice.
Afin d’y remédier, des versions améliorées de CRISPR ont été proposées, permettant par exemple de modifier directement les séquences cibles plutôt que s’appuyer sur les cycles de réparation-mutation. Certaines permettent de modifier les bases nucléotidiques sans passer par le processus de clivage, tandis que d’autres permettent de convertir les ARNg en ADN et de les insérer au niveau de la séquence à cibler. Cependant, là encore, la précision reste limitée.
Une équipe de l’Arc Institute, de l’Université de Californie à Berkeley, de Stanford et de Tokyo, propose une nouvelle technique d’édition programmable permettant d’insérer directement de nouvelles séquences d’ADN dans le génome en utilisant des « ARN ponts » – une nouvelle catégorie d’ARN guides. Cela permettrait d’améliorer considérablement la précision de l’édition génétique à l’aide d’une seule séquence d’insertion.
« Le système de pont ARN est un mécanisme fondamentalement nouveau pour la conception du génome », explique dans un article de blog de l’Arc Institute le chercheur principal de l’étude, Patrick Hsu, également professeur de bio-ingénierie à l’Université de Californie à Berkeley. « La recombinaison de pont peut modifier universellement le matériel génétique par l’insertion, l’excision, l’inversion de séquences spécifiques et plus encore, permettant ainsi une édition du génome vivant plus efficace que CRISPR ».
« L’ARN pont » : un adaptateur universel permettant de cibler n’importe quelle partie du génome
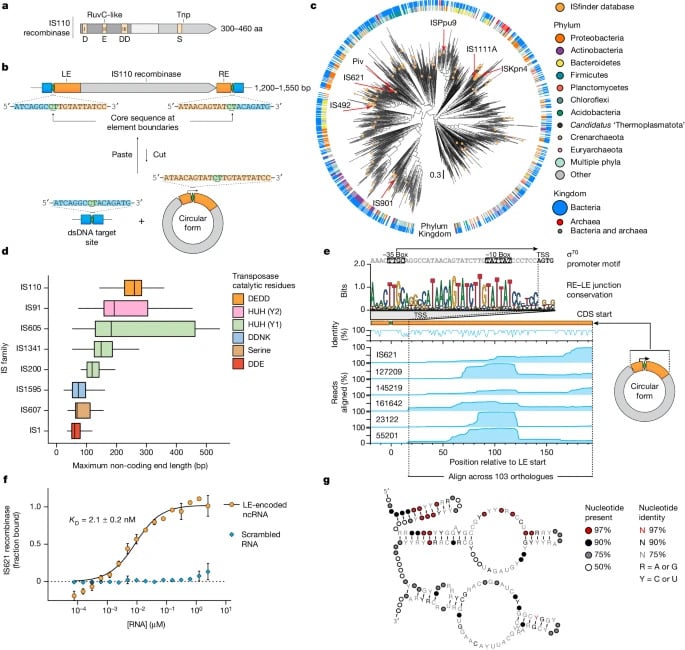
Découvert chez les bactéries et les archées, le nouveau système d’édition moléculaire étudié par l’équipe est basé sur une séquence dite « d’insertion 110 (IS110) ». Il fait partie d’un grand ensemble de séquences transposables (ou « gènes sauteurs ») qui se coupent, se déplacent à l’intérieur du génome, puis se collent (ou s’insèrent) entre deux brins spécifiques.
Ces courtes séquences sont présentes au niveau des cellules de tous les êtres vivants et ont évolué en devenant de véritables outils de manipulation de l’ADN. L’IS110 en particulier est constitué d’un gène codant pour une enzyme recombinase, responsable de la recombinaison génétique. Il s’agit d’un processus qui conduit à la modification de l’association physique entre deux segments d’ADN.
Les experts de la nouvelle étude — publiée dans la revue Nature — ont utilisé la cryomicroscopie électronique pour étudier les structures moléculaires du complexe ARN pont-recombinase. Ils ont progressé par étapes depuis le processus de clivage de l’IS110 jusqu’à la phase de recombinaison.
Ils ont découvert que lorsque l’IS110 s’excise du génome, les extrémités non codantes du brin d’ADN se rejoignent pour produire deux boucles d’ARN pont. L’une des boucles se lie à l’IS110 tandis que l’autre se lie au brin cible au niveau duquel la nouvelle séquence sera insérée — ce qui fait de l’ARN pont le premier exemple d’ARNg bispécifique. La recombinase induit ensuite le processus d’insertion.
D’autre part, chaque ARN pont est programmable, ce qui permet d’associer n’importe quelle séquence d’ADN cible et donneuse. En d’autres termes, l’outil permet d’insérer n’importe quelle séquence d’ADN sur n’importe quel emplacement génomique cible. « Ces ponts ARN programmables distinguent IS110 des autres recombinases connues, qui ne contiennent pas de composant ARN et ne peuvent pas être programmées », explique le coauteur principal de l’étude, Nicolas Perry, également de l’Arc Institute et de l’Université de Californie. Pour l’analogie, « c’est comme si l’ARN pont était un adaptateur secteur universel qui rend IS110 compatible avec n’importe quelle prise », indique-t-il.


Une précision de plus de 94 %
Ce mécanisme confère au nouveau système un niveau de précision qui ne pourrait jamais être obtenu avec la technique CRISPR. Lors d’essais sur des bactéries Escherichia coli, l’équipe a démontré que l’efficacité d’insertion d’un gène donné était de plus de 60 %, tandis que la capacité à s’insérer au niveau du bon emplacement était supérieure à 94 %.
Par ailleurs, la technique permet de recombiner deux brins d’ADN sans libérer les extrémités précédemment clivées de ces derniers. Ces séquences dites « cicatrices » constituent une limite non négligeable des techniques d’édition génétique utilisées actuellement.
Toutefois, il est important de noter que la technique n’a pour le moment été éprouvée qu’au niveau des bactéries et son efficacité sur d’autres types de cellules, y compris humaines, reste à prouver. Néanmoins, elle pourrait à terme ouvrir la voie à de nouveaux types de thérapies géniques. Selon les experts, les prochaines recherches s’orienteront sur l’application de l’outil sur les cellules humaines, l’amélioration de sa précision et de son efficacité, ainsi que l’exploration de potentielles fonctions supplémentaires.
Vidéo explicative de l’étude :