PlantRNA-FM, un nouveau modèle d’IA formé sur les données d’ARN de plus de 1 100 espèces végétales, peut déchiffrer le « langage » génétique des plantes. Le modèle a appris à décoder l’agencement des motifs d’ARN et à en identifier les fonctions spécifiques, un peu de la même manière qu’il décoderait l’agencement des lettres de l’alphabet pour comprendre le langage humain. La technologie permettrait, à terme, de développer des techniques d’amélioration de cultures ou de conservation des espèces végétales.
Le transcriptome, l’ensemble des ARN issus de la transcription du génome, rassemble un large éventail de motifs ayant un impact sur diverses fonctions biologiques clés, telles que la traduction des protéines. Des études ont montré que les caractéristiques et la structure de ces motifs sont essentielles aux processus biologiques. Chez les plantes par exemple, les températures fraîches (environ 20 °C) favorisent le repliement de certains motifs d’ARN, probablement liés à des processus adaptatifs.
Cependant, l’identification des motifs d’ARN fonctionnels dans les transcriptomes constitue un défi de taille en raison des dizaines de milliers de combinaisons possibles de bases nucléotidiques qui les composent. Pour une séquence de 50 nucléotides, par exemple, le nombre de combinaisons possibles serait de l’ordre de 1,27 × 1030.
Il a été suggéré que les modèles de fondation (Foundation model ou FM) pourraient permettre d’identifier et de décoder ces motifs. Il s’agit de modèles d’IA se distinguant par leur grande taille (jusqu’à plusieurs milliards de paramètres) et pouvant être pré-entraînés de manière auto-supervisée sur divers types de données non étiquetées – ce qui les rend particulièrement adaptés à la biologie.
Les FM sont également très adaptables et peuvent être configurés pour analyser des processus biologiques complexes. À l’instar des lettres de l’alphabet qui sont assemblées pour former des mots et des phrases, les nucléotides composant les motifs d’ARN sont agencés selon leurs fonctions spécifiques. Les FM pourraient ainsi les décoder de la même manière qu’un langage humain.
Développé par des chercheurs du John Innes Centre, de l’Université d’Exeter, de la Northeast Normal University et de l’Académie chinoise des sciences, PlantRNA-FM est le premier modèle spécifiquement conçu pour identifier les motifs fonctionnels d’ARN chez les plantes. « Cette avancée ouvre de nouvelles possibilités pour mieux comprendre et modifier les plantes, avec des implications potentielles pour l’amélioration des cultures et la conception génétique assistée par IA », a déclaré dans un communiqué Yiliang Ding, du Département de biologie cellulaire et du développement du John Innes Centre, co-auteur de la nouvelle étude.

Une technique similaire au décodage du langage humain
Afin de décoder l’ARN, de précédents FM ont été pré-entraînés à l’aide d’un grand nombre de séquences et ont démontré d’excellentes performances dans la conception de nouvelles séquences. Cependant, les informations basées uniquement sur les séquences sont limitées, car l’ARN peut former des motifs de structure secondaires ou tertiaires assurant des fonctions spécifiques. Les chercheurs de cette nouvelle étude suggèrent qu’il est essentiel de développer un FM intégrant à la fois des informations sur les séquences et la structure de l’ARN, pour faciliter l’identification des motifs fonctionnels.
Dans cette vision, PlantRNA-FM a été entraîné avec un ensemble de 54 milliards d’éléments d’informations d’ARN, englobant les transcriptomes de 1124 espèces végétales du monde entier. « En incorporant des séquences d’ARN, des annotations et des informations sur la structure de 1 124 espèces végétales distinctes, PlantRNA-FM capture la grande diversité des transcriptomes végétaux », expliquent les chercheurs dans leur document, publié dans la revue Nature Machine Intelligence.
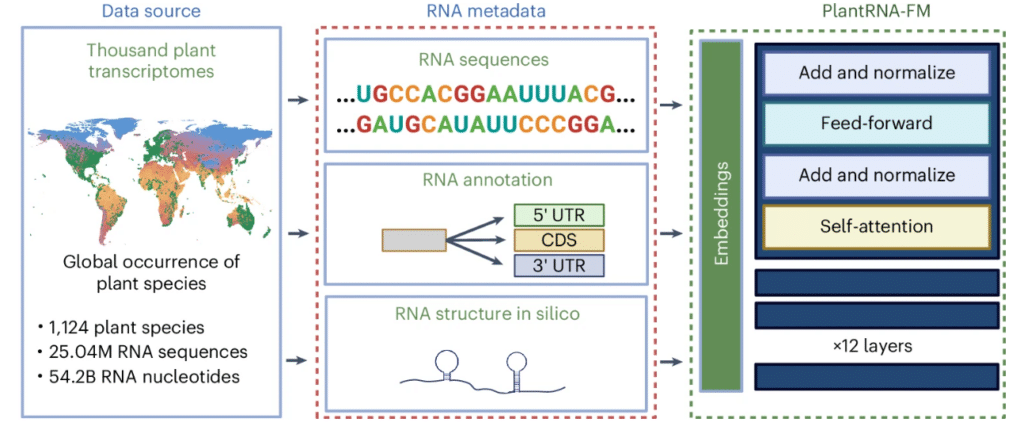


De la même manière qu’il décoderait les phrases d’un langage humain, le modèle a appris à comprendre la logique dans l’agencement des séquences et des structures d’ARN. Selon l’équipe, il est parvenu à effectuer des prédictions précises sur les fonctions des séquences et à identifier les motifs fonctionnels spécifiques au sein des transcriptomes. Ces prédictions ont été confirmées par des expériences montrant que les motifs d’ARN identifiés par PlantRNA-FM jouent un rôle dans l’efficacité de la traduction de l’information génétique en protéines.
« Alors que les séquences d’ARN peuvent paraître aléatoires à l’œil humain, notre modèle d’IA a appris à décoder les modèles cachés en leur sein », explique Haopeng Yu, chercheur postdoctoral au John Innes Centre et également coauteur de l’étude. Cette technologie pourrait contribuer à une meilleure compréhension du monde végétal et offrir des pistes pour répondre à des défis mondiaux comme l’autosuffisance alimentaire et la résilience climatique des cultures de subsistance et des espèces menacées. Selon les chercheurs, le modèle pourrait aussi être appliqué à l’étude des invertébrés et des bactéries, ce qui pourrait potentiellement ouvrir la voie à de grandes avancées dans la compréhension du fonctionnement des écosystèmes.


