En utilisant LucaProt, un nouvel algorithme d’apprentissage profond, des chercheurs ont identifié 161 979 espèces de virus à ARN « cachées », dont 70 458 jusqu’à présent inconnues de la science. Les génomes de la majorité de ces espèces virales, dont les séquences sont surnommées « séquences matière noire », avaient déjà été séquencés, mais ils sont si étranges qu’ils n’avaient jusqu’ici jamais pu être identifiés. Beaucoup d’entre eux vivent en milieux extrêmes, témoignant d’une remarquable biodiversité.
Les virus sont omniprésents sur la planète et infectent un large éventail d’animaux, de plantes et de bactéries. Ils jouent un rôle essentiel dans l’équilibre des écosystèmes en régulant la population de leurs espèces hôtes. L’identification et la caractérisation des virus sont généralement basées sur l’analyse d’ARN polymérase dépendante de l’ARN (RdRP), un composant spécifique des génomes des virus à ARN. Cela a permis, il y a quelques années, d’identifier des dizaines de milliers d’espèces virales, étendant d’au moins 10 fois la « virosphère » connue.
Cependant, malgré les avancées en matière de séquençages métagénomiques, seule une petite portion des virus à ARN a été identifiée à ce jour, tandis que de nombreux groupes largement répandus sont probablement non identifiés. Selon Artem Babaian, de l’Université de Toronto (au Canada), il y aurait « un puits sans fond » de virus non identifiés. Cela est en partie dû au fait que les techniques métagénomiques standard ne permettent pas d’identifier les RdRP très divergents.
Surnommées « séquences matière noire », ces séquences « cachées » seraient nombreuses et difficiles à identifier. En outre, de nouvelles techniques doivent être mises au point pour les caractériser avec précision. Certains virus pourraient infecter l’humain et les caractériser permettrait peut-être d’expliquer certaines maladies mystérieuses. Une potentielle implication virale a par exemple récemment été proposée pour Alzheimer, dont l’étiologie exacte fait encore aujourd’hui l’objet de débats.
Lucaprot a été développé à cet effet par des chercheurs de l’Université Sun Yat-sen (en Chine) et de l’Université de Sydney (en Australie). « Notre méthode d’IA a pu organiser et catégoriser toutes ces informations disparates, mettant en lumière pour la première fois la signification de cette ‘matière noire’ », explique dans un communiqué de l’Université de Sydney Edward Holmes, coauteur de l’étude — publiée dans la revue Cell.
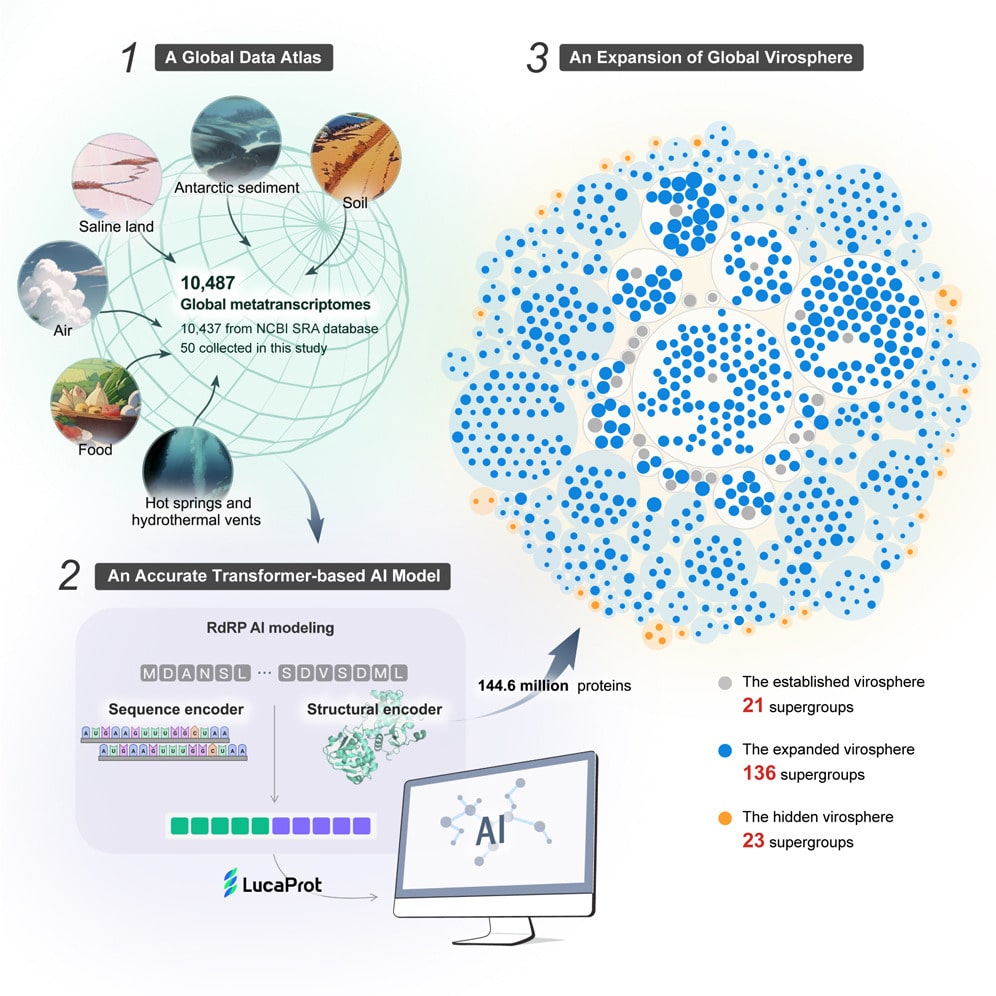
D’étranges virus vivant en milieux extrêmes
Pour l’identification de séquences virales, les algorithmes d’apprentissage profond possèdent plusieurs avantages par rapport aux approches bioinformatiques conventionnelles, notamment une précision accrue, la prise en charge de très grands volumes de données en un temps record et la capacité d’auto-apprentissage. Selon Mang Shi de l’Université Sun Yat-sen, également coauteur de l’étude : « nous avions l’habitude de nous appuyer sur des pipelines bioinformatiques fastidieux pour la découverte de virus, ce qui limitait la diversité que nous pouvions explorer ».
Lucaprot est basé sur les transformeurs, l’une des architectures de modèles d’apprentissage profond les plus efficaces pour effectuer des prédictions de données. Contrairement aux réseaux neuronaux récurrents, ils permettent de traiter les données dans un ordre aléatoire, permettant ainsi de réduire considérablement le temps d’entraînement. L’algorithme est alimenté par les données de séquençage et de prédiction de protéines d’ESMFold, un outil d’IA développé par Meta. Il a ensuite été entraîné à reconnaître les RdRps viraux et les « séquences matière noire ».
À noter que des algorithmes d’apprentissage profond ont précédemment été utilisés pour l’identification de virus à partir de données génomiques et métagénomiques. Cependant, leurs architectures étaient généralement basées soit sur les réseaux neuronaux convolutionnels (CNN), soit sur les réseaux neuronaux récurrents. La première rencontre des difficultés avec les séquences de longueur variable, tandis que la seconde peine à traiter spécifiquement de longues séquences — limitant ainsi leur capacité à identifier les séquences divergentes.
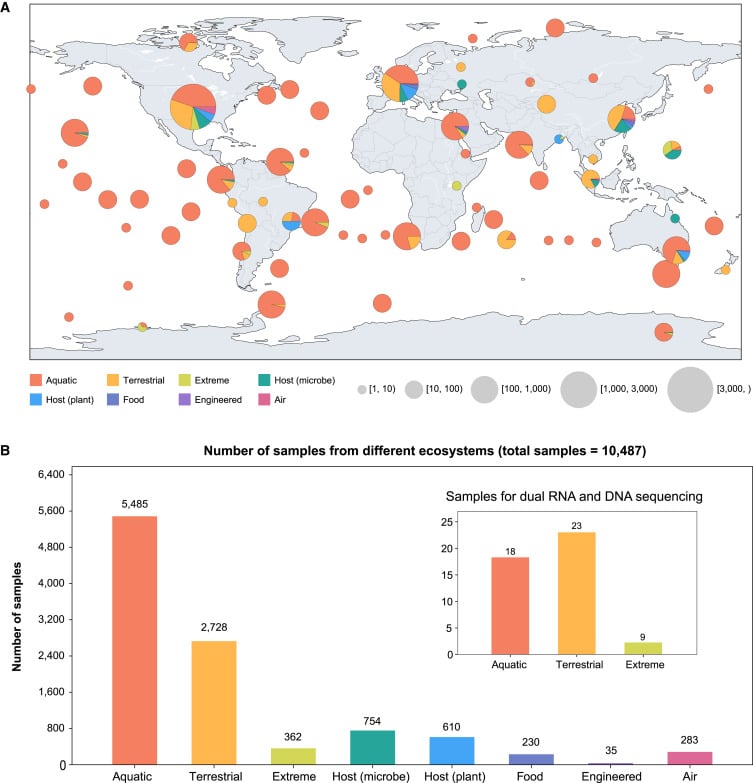
Lucaport a identifié au total 161 979 espèces et 180 supergroupes de virus à ARN. 70 458 appartiennent à des espèces jusqu’ici inconnues, certaines à séquences étranges et exceptionnellement longues (jusqu’à 47 250 nucléotides). Ces nouvelles espèces sont en outre présentes dans des environnements incroyablement diversifiés, allant de l’air aux sources hydrothermales en passant par les lacs salés. Leur diversité ainsi que leur densité varient considérablement en fonction des écosystèmes.
Il s’agirait du plus grand nombre de virus identifiés en une seule étude. Leur identification améliorerait considérablement la compréhension de la biodiversité virale dans la biosphère. « Le fait que les environnements extrêmes abritent autant de types de virus n’est qu’un autre exemple de leur diversité phénoménale et de leur ténacité à vivre dans les environnements les plus difficiles, nous donnant potentiellement des indices sur la façon dont les virus et d’autres formes de vie élémentaires sont apparus », suggère Holmes.
Des millions d’autres espèces à découvrir
Par ailleurs, malgré le nombre d’espèces virales identifiées, l’équipe estime que l’étude n’a fait qu’effleurer la surface et des millions d’autres espèces restent encore à découvrir. La prochaine étape de la recherche consistera donc à utiliser Lucaport pour identifier d’autres groupes viraux. L’approche pourrait aussi être appliquée à l’identification des bactéries et des parasites.
D’autre part, les hôtes potentiels des virus nouvellement répertoriés n’ont pas encore été identifiés. Shi et ses collègues développent actuellement un nouveau modèle d’IA dans ce sens et espèrent éclairer davantage le rôle de ces virus dans leurs niches environnementales. Des travaux seront également réalisés afin de déterminer si certains de ces virus peuvent infecter les archées, des organismes eucaryotes pour lesquels aucun virus à ARN connu n’a été identifié comme infectant.