La biologie synthétique est un domaine de recherche en pleine expansion depuis ces dernières années. Le séquençage génétique et la possibilité de synthétiser du matériel biologique offrent aux chercheurs un tout nouveau cadre expérimental pour le traitement de maladies génétiques ou la meilleure compréhension de certains mécanismes génétiques. Récemment, une entreprise de biotechnologies a présenté une nouvelle méthode de synthèse d’ADN qui pourrait révolutionner le domaine.
Avec l’essor du séquence génétique, les scientifiques peuvent aujourd’hui pouvoir cartographier le génome plus vite que jamais. Toutefois, si la lecture d’ADN semble relativement aisée, « l’écriture » d’ADN est, quant à elle, bien plus complexe. Jusqu’à présent, seuls de très petites séquences ont été synthétisées artificiellement via un processus biochimique lent et coûteux.
Mais cette situation problématique est sur le point de changer. Une entreprise de biotechnologies française a annoncé, lors d’une conférence sur la biologie synthétique à San Francisco, qu’en utilisant une nouvelle méthode basée sur des enzymes similaires de synthèse de l’ADN chez les organismes vivants (les ADN polymérases), il était possible de synthétiser une séquence longue de 150 nucléotides. Ce nombre brise le record de 50 nucléotides annoncé quelques mois auparavant par un autre laboratoire.
Sur le même sujet :
« C’est une véritable étape clé » affirme George Church, généticien à l’université de médecine d’Harvard. Selon lui, ces résultats placent la synthèse enzymatique d’ADN sur une courbe de croissance exponentielle. Et si cette technique fonctionne efficacement, les chercheurs souhaitant créer de nouveaux génomes ou analyser des mécanismes génétiques précis, auront bientôt de plus longues séquences d’ADN synthétisées, plus rapidement et à moindres coûts.
Bien que la synthèse chimique d’ADN usuelle ait été miniaturisée et automatisée, le processus sous-jacent, appelé chimie phosphoramidite, est le même depuis son apparition dans les années 1980. Il implique l’ajout de nucléotides les uns après les autres, individuellement protégés par un groupe terminal bloquant toute interaction chimique afin d’étendre la séquence de manière stable, le groupe étant enlevé lors de l’ajout du nucléotide suivant.
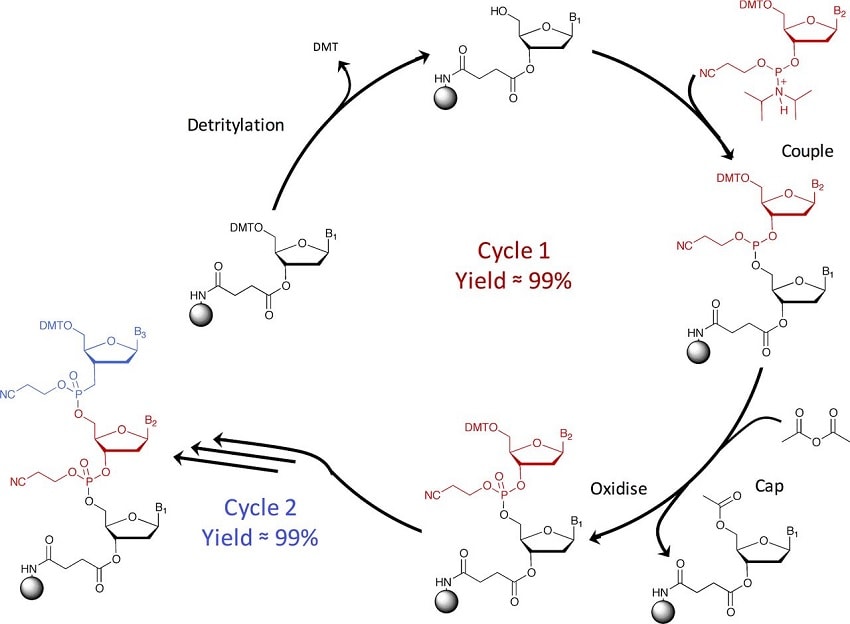
Cependant, cette technique présente plusieurs problèmes. Pour chaque nucléotide ajouté, la probabilité d’erreur génétique est de 0.5%. Plus la séquence est longue, plus les probabilités de contenir des erreurs sont élevées, limitant virtuellement la séquence à 300 nucléotides. Par conséquent, les chercheurs souhaitant travailler sur des gènes contenant des milliers de bases doivent laborieusement fixer chaque séquence de 300 nucléotides les unes aux autres.
La synthèse enzymatique, quant à elle, promet un processus plus simple et efficace. Elle se base en effet sur l’ADN polymérase, une enzyme impliquée dans la synthèse d’ADN chez les cellules vivantes. L’ADN polymérase possède une vitesse de synthèse élevée tout en supprimant presque totalement tout risque d’erreur.
Bien que la synthèse enzymatique d’ADN ne soit apparue qu’au début des années 2010, environ une demi-douzaine d’entreprises travaillent déjà sur cette technique, tandis que d’autres travaillent sur des méthodes de stockage génétique de données et d’analyses biomédicales, pour quand la synthèse enzymatique sera prête. Selon Michael Kamdar, PDG de Molecular Assemblies, une entreprise de synthèse enzymatique d’ADN située en Californie, le marché de la synthèse chimique d’ADN classique représente environ un milliard de dollars par an.
Le marché du stockage de données représente quant à lui environ 14 milliards de dollars, bien que l’ADN n’y contribue que pour une très petite fraction. « D’ici 2 à 3 ans, vous pourrez voir des applications de la synthèse enzymatique d’ADN sur le marché, que nous en soyons à l’origine ou non » explique Sylvain Gariel, co-fondateur de l’entreprise française ADN Script.
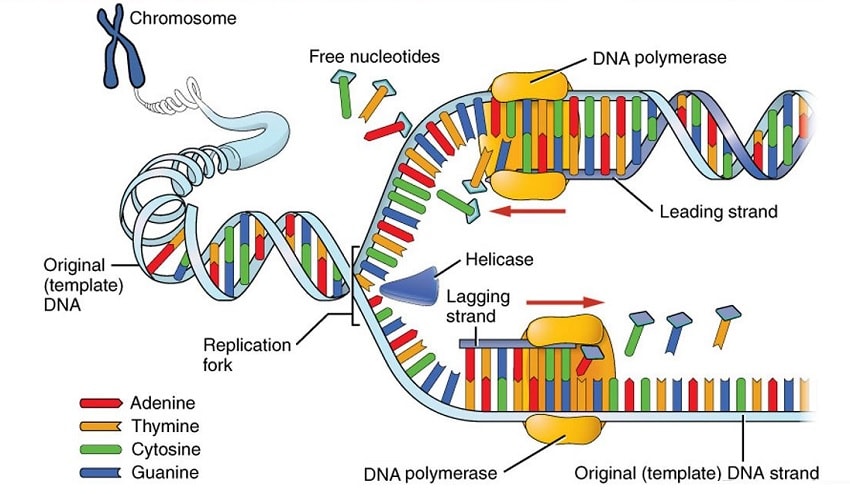


Passer de la synthèse chimique à la synthèse enzymatique a représenté un défi pour les chercheurs. Dans les cellules vivantes, l’ADN polymérase se sert d’un brin d’ADN modèle pour synthétiser le brin complémentaire, assemblant les paires A-T et C-G. Une polymérase spécifique présente dans les cellules immunitaires, appelée déxorybonucléotidyl transférase terminale (TdT), fonctionne sans brin modèle, et constitue donc la candidate idéale pour la synthèse enzymatique d’ADN.
Puisque la TdT ajoute des nucléotides aléatoirement, les scientifiques ont dû chercher un moyen de lui faire suivre une séquence déterminée. Pour ce faire, Gariel ajoute une protection sur chaque nucléotide qui, comme dans la synthèse chimique, empêche la TdT d’ajouter plus d’un nucléotide à la fois. Une fois la base désirée ajoutée, sa protection est enlevée et le cycle se répète. Selon Gariel, ajouter chaque nucléotide ne nécessite que 5 minutes, avec une précision de 99.5%.
La plupart des experts estiment que les applications de la synthèse enzymatique d’ADN sont nombreuses. « Le potentiel de la synthèse enzymatique surpasse celui de la synthèse chimique » indique Kamdar. Le PDG d’ADN Script, Thomas Ybert, espère pouvoir produire des séquences de 1000 bases quotidiennement. L’entreprise ambitionne également, d’ici 2020, de commercialiser des synthétiseurs enzymatiques automatiques d’ADN.
Une telle avancée pourrait réduire les coûts de synthèse d’ADN de un ou deux ordres de grandeurs. Cela sera plus facile et moins cher pour les biologistes de synthétiser et tester de nouveaux gènes, que ce soit pour des applications technologiques ou la thérapie génétique. Cela pourrait également révolutionner le stockage de données. En effet, théoriquement, toutes les données actuelles du monde pourraient tenir dans une séquence génétique de taille inférieure à celle d’une petite mallette.



