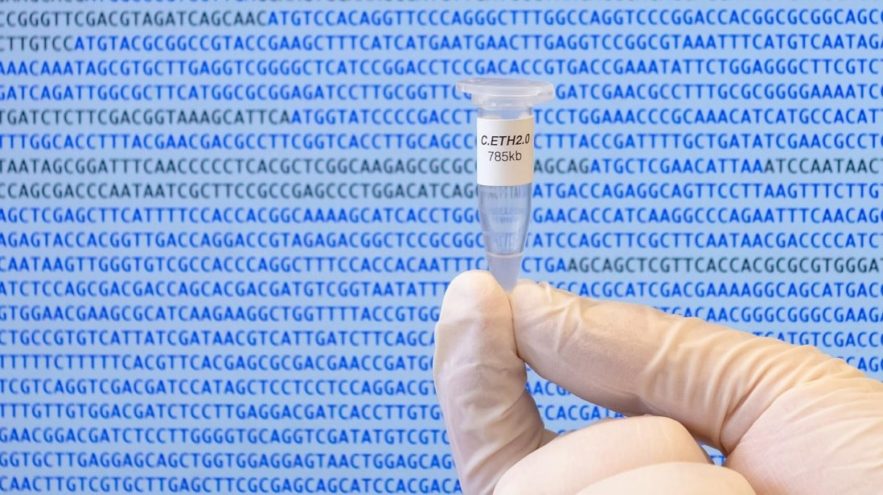
Toutes les séquences génétiques d’organismes connues dans le monde entier sont stockées dans une base de données appartenant au National Center for Biotechnology Information des États-Unis. Aujourd’hui, la base de données comporte une entrée supplémentaire : Caulobacter ethensis-2.0. Il s’agit du premier génome d’un organisme vivant entièrement généré par ordinateur, mis au point par des scientifiques de l’ETH Zürich, en Suisse.
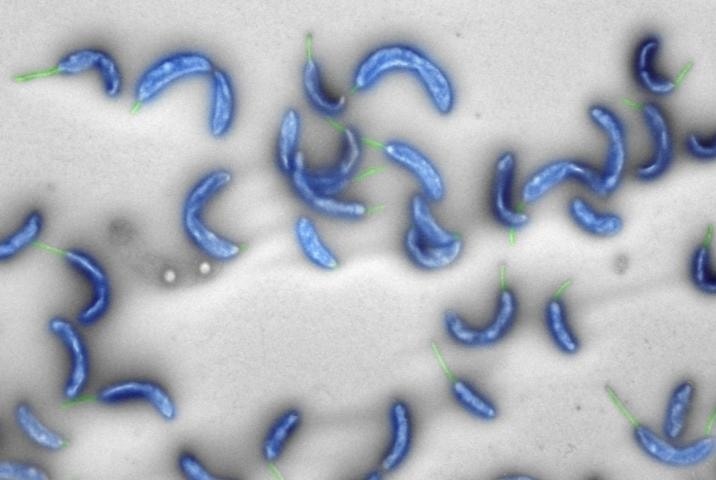
Cependant, il convient de souligner que, bien que le génome de C. ethensis-2.0 ait été produit physiquement sous la forme d’une très grande molécule d’ADN, il n’existe pas encore d’organisme correspondant dans la nature. C. ethensis-2.0 est basé sur le génome d’une bactérie d’eau douce bien étudiée et inoffensive, Caulobacter crescentus, une bactérie naturellement présente dans les eaux de source, les rivières et les lacs du monde entier.
Elle ne provoque aucune maladie. C. crescentus est également un organisme modèle couramment utilisé dans les laboratoires de recherche pour étudier la vie des bactéries. Le génome de cette bactérie contient 4000 gènes. Les scientifiques ont précédemment démontré que seuls environ 680 de ces gènes sont essentiels à la survie de l’espèce en laboratoire. Les bactéries avec ce génome minimal sont viables en laboratoire.
Beat Christen, professeur de biologie des systèmes expérimentaux à l’ETH Zürich et son frère Matthias Christen, chimiste à l’ETH Zürich, ont pris comme point de départ le génome minimal de C. crescentus. Ils ont entrepris de synthétiser chimiquement ce génome à partir de rien, sous la forme d’un chromosome en forme d’anneau continu. Les résultats de l’expérience ont été publiés dans la revue PNAS.
Une telle tâche était auparavant considérée comme un véritable tour de force : le génome bactérien synthétisé chimiquement présenté il y a onze ans par le pionnier américain de la génétique, Craig Venter, était le résultat de dix années de travail de vingt scientifiques. Le coût du projet se serait élevé à 40 millions de dollars.
Perfectionner la technologie de synthèse génomique
Alors que l’équipe de Venter réalisait une copie exacte d’un génome naturel, les chercheurs de l’ETH Zürich ont radicalement modifié leur génome en utilisant un algorithme informatique. Leur motivation était double : premièrement, rendre la production de génomes beaucoup plus facile et deuxièmement, répondre aux questions fondamentales de la biologie.
Pour créer une molécule d’ADN de la taille d’un génome bactérien, les scientifiques doivent procéder étape par étape. Dans le cas du génome de Caulobacter, les scientifiques de l’ETH Zürich ont synthétisé 236 segments du génome, qu’ils ont ensuite reconstitués.
« La synthèse de ces segments n’est pas toujours facile » explique Matthias Christen. « Les molécules d’ADN possèdent non seulement la capacité de coller à d’autres molécules d’ADN, mais en fonction de la séquence, elles peuvent également se tordre en boucles et en nœuds, ce qui peut entraver le processus de production ou rendre la fabrication impossible ».
Simplifier la séquence ADN pour une synthèse optimale
Pour synthétiser les segments du génome de la manière la plus simple possible, puis rassembler tous les segments, les scientifiques ont radicalement simplifié la séquence du génome sans modifier les informations génétiques réelles. Il y a beaucoup de latitude pour la simplification des génomes, car la biologie comporte des redondances intégrées pour stocker les informations génétiques. Par exemple, pour de nombreux composants protéiques (acides aminés), il existe deux, quatre possibilités voire plus d’écrire leurs informations dans l’ADN.
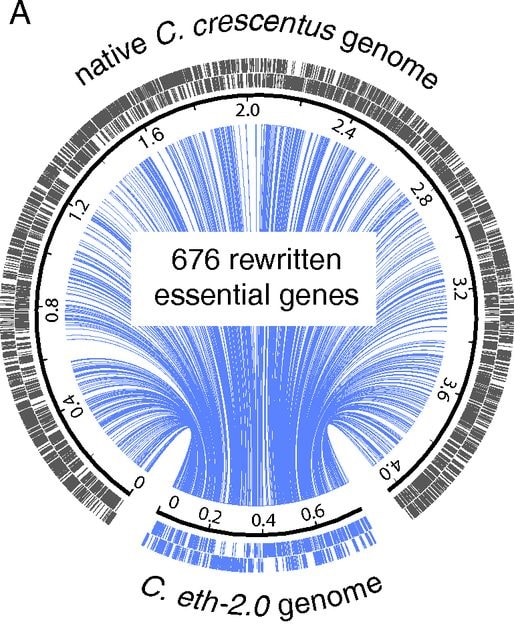
L’algorithme développé par les scientifiques exploite de manière optimale cette redondance du code génétique. À l’aide de cet algorithme, les chercheurs ont calculé la séquence d’ADN idéale pour la synthèse et la construction du génome, qu’ils ont finalement utilisée pour leurs travaux.
En conséquence, les scientifiques ont introduit de nombreuses petites modifications dans le génome minimal, qui sont toutefois impressionnantes dans leur intégralité : plus d’un sixième des 800’000 lettres d’ADN du génome artificiel ont été remplacées, par rapport au système minimal initial.
« Grâce à notre algorithme, nous avons complètement réécrit notre génome dans une nouvelle séquence de lettres ADN qui ne ressemble plus à la séquence d’origine. Cependant, la fonction biologique au niveau de la protéine reste la même » explique Beat Christen.
Mieux comprendre le génome et ses fonctionnalités
Le génome réécrit est également intéressant d’un point de vue biologique. « Notre méthode est un test décisif pour voir si nous, les biologistes, avons bien compris la génétique, et elle nous permet de mettre en évidence d’éventuelles lacunes dans nos connaissances » ajoute Christen.
Naturellement, le génome réécrit ne peut contenir que des informations que les chercheurs ont réellement comprises. Les informations supplémentaires cachées éventuelles qui se trouvent dans la séquence d’ADN et n’ont pas encore été comprises par les scientifiques auraient été perdues lors de la création du nouveau code.
Sur le même sujet : Pour la toute première fois, des scientifiques ont créé un ADN synthétique avec 4 lettres supplémentaires
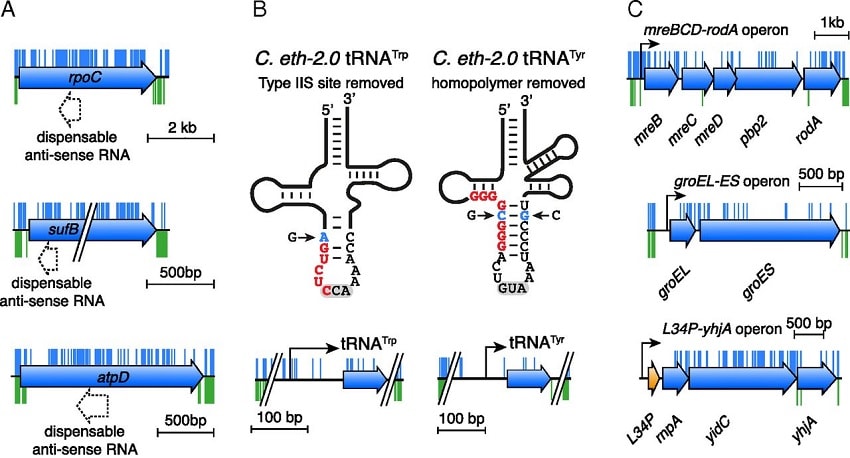
À des fins de recherche, les scientifiques ont produit des souches de bactéries contenant à la fois le génome naturel de Caulobacter et des segments du nouveau génome artificiel. En désactivant certains gènes naturels chez ces bactéries, les chercheurs ont pu tester les fonctions des gènes artificiels. Ils ont testé chacun des gènes artificiels dans un processus en plusieurs étapes.
Dans ces expériences, les chercheurs ont découvert que seulement environ 580 des 680 gènes artificiels étaient fonctionnels. « Grâce aux connaissances que nous avons acquises, il nous sera toutefois possible d’améliorer notre algorithme et de développer une version 3.0 du génome, entièrement fonctionnelle » déclare Christen.
Des applications biotechnologiques à fort potentiel
« Même si la version actuelle du génome n’est pas encore parfaite, nos travaux montrent néanmoins que les systèmes biologiques sont construits de manière si simple qu’à l’avenir, nous pourrons élaborer les spécifications de conception sur ordinateur en fonction de nos objectifs, et ensuite les construire » déclare Matthias Christen.
Et cela peut être accompli de manière relativement simple, comme le souligne Beat Christen : « Ce qui a pris dix ans avec l’approche de Craig Venter, notre petit groupe l’a réalisé avec notre nouvelle technologie dans un délai d’un an, avec des coûts de fabrication de 120’000 francs suisses (environ 108’000 Euros). Nous pensons qu’il sera bientôt possible de produire des cellules bactériennes fonctionnelles avec un tel génome ».
Un tel développement aurait un grand potentiel. Parmi les applications futures possibles figurent les micro-organismes synthétiques, qui pourraient être utilisés en biotechnologie pour la production de molécules complexes pharmaceutiquement actives ou de vitamines, par exemple. La technologie peut être utilisée universellement pour tous les micro-organismes, pas seulement pour Caulobacter. Une autre possibilité serait la production de vaccins à ADN.
« Aussi prometteurs que puissent être les résultats de la recherche et les applications possibles, ils exigent une discussion en profondeur de la société sur les objectifs pour lesquels cette technologie peut être utilisée et, parallèlement, sur la manière dont les abus peuvent être évités » déclare Christen.
On ne sait toujours pas quand la première bactérie dotée d’un génome artificiel sera produite — mais il est maintenant clair qu’elle peut être développée, et qu’elle le sera. « Nous devons utiliser le temps dont nous disposons pour des discussions intensives entre scientifiques, ainsi que dans l’ensemble de la société. Nous sommes prêts à contribuer à cette discussion grâce à tout le savoir-faire que nous possédons » conclut le chercheur.