Les différences individuelles sont nombreuses en matière de santé, par exemple en ce qui concerne les effets secondaires des médicaments. Mais à quel point sommes-nous différents ? Pour la première fois, un laboratoire a utilisé des tubes digestifs humains entiers provenant de trois donneurs d’organes pour montrer comment les types de cellules diffèrent dans toutes les régions de l’intestin, mettre en lumière les fonctions cellulaires et montrer les différences d’expression génétique entre ces cellules et entre les individus.
Appelés « intestin », le côlon et l’intestin grêle sont des organes distincts dont les rôles sont uniques. La lumière intestinale (l’intérieur des intestins) est tapissée de cellules épithéliales (souches et différenciées) qui se renouvellent chaque semaine, et dont les rôles sont nombreux. « Notre laboratoire a montré qu’il était possible de connaître la fonction de chaque type de cellule dans des processus importants, tels que l’absorption des nutriments, la protection contre les parasites, la production de mucus et d’hormones qui régulent le comportement alimentaire et la motilité intestinale », a déclaré dans un communiqué Scott T. Magness, professeur au département d’ingénierie biomédicale UNC-NC State et auteur principal de l’étude.
Si les fonctions physiologiques varient le long de l’intestin, la manière dont les lignées diffèrent sur l’axe intestin grêle-côlon était mal connue. De plus, il y a peu de donneurs pour certaines régions de l’intestin et l’analyse épithéliale est ainsi limitée. « Une telle exploration reviendrait à regarder les États-Unis depuis l’espace, mais à n’étudier que ce qui se passe dans le Massachusetts, l’Oklahoma et la Californie », a continué Magness.
Jusqu’à présent, les chercheurs utilisaient des biopsies pour analyser les caractéristiques générales des types de cellules épithéliales. Dans la nouvelle étude, ils ont prélevé des milliers de cellules individuelles à la fois dans l’intestin grêle et dans le côlon, et ils ont ensuite étudié les rôles potentiels de ces cellules, à l’aide des gènes exprimés.
12 590 cellules épithéliales analysées
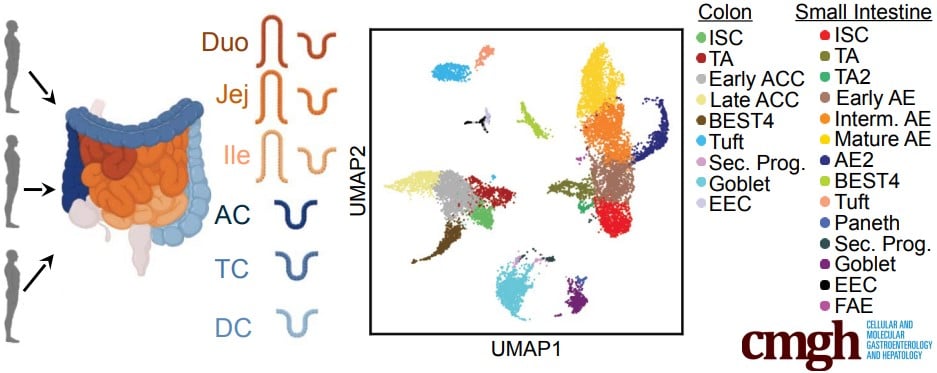
Les chercheurs ont procédé à une analyse complète de l’épithélium de l’intestin humain adulte (comprenant le duodénum, le jéjunum, l’iléon et le côlon ascendant, transverse et descendant) en utilisant les organes de trois donneurs. Dans un premier temps, ils ont prélevé la couche épithéliale des tracts intestinaux reçus. Ils ont ensuite utilisé des enzymes pour décomposer l’épithélium en cellules individuelles.
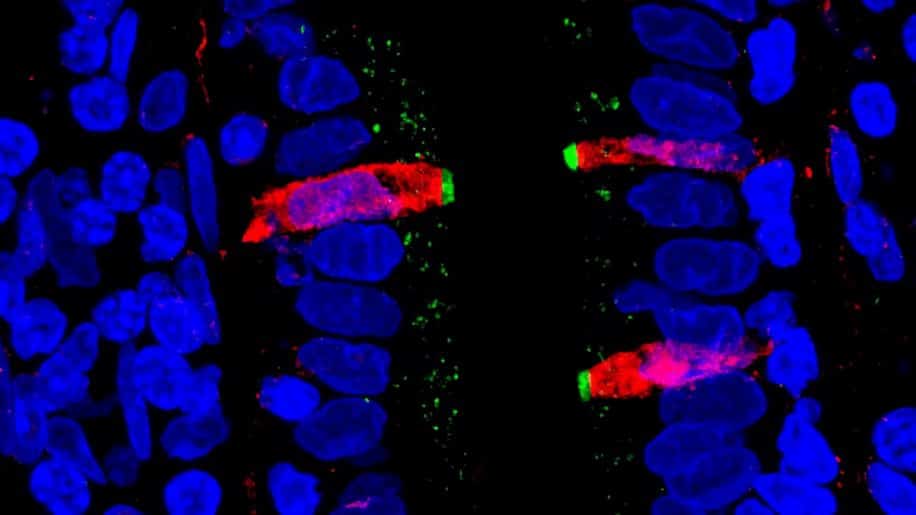
Au total, ce ne sont pas moins de 12 590 cellules épithéliales uniques provenant de donneurs traités de manière indépendante qui ont été évaluées pour former un « atlas de cellules ». L’équipe a sondé des lignées cellulaires peu étudiées, comme les cellules de Paneth (PCs), les cellules BEST4+ et l’épithélium associé aux follicules.
Joseph Burclaff, l’un des auteurs de l’étude, a ajouté : « Si vous regardez le mucus intestinal, qui est un mélange complexe qui protège les cellules, nous montrons quelles cellules expriment diverses protéines de mucine, en quelle quantité, et dans quelles régions du tube digestif. Nous avons aussi examiné où sont exprimées les enzymes spécifiques qui digèrent les aliments ».
La méthode de transcriptomique — ou séquençage — à cellule unique a permis d’obtenir une résolution sans précédent de la fonction tissulaire au niveau cellulaire. Cette technique donne lieu à l’identification de gènes actifs dans chaque type de cellule — ce qui représente une très grande quantité de données — et a permis de découvrir comment ces différences génétiques modifient l’identité et la fonction cellulaires.
Les cellules ont été assignées à 25 groupes de lignées épithéliales. Les cellules BEST4+ présentent des différences de maturation entre l’intestin grêle et le côlon et les cellules de Paneth possèdent une large capacité d’interaction avec le système immunitaire. L’expression différentielle des récepteurs et des cibles médicamenteuses entre les lignées révèle une variation biologique et un potentiel de réponse variés.
Certains effets secondaires gastro-intestinaux seraient dus à l’expression de gènes des cellules épithéliales
Les chercheurs ont ainsi pu observer les différences entre les types de cellules dans l’ensemble du tube digestif, et même différents niveaux d’expression génétique dans les mêmes types de cellules issues de trois personnes différentes. Une avancée pour comprendre pourquoi certaines personnes (et pas d’autres) présentent une toxicité à certains médicaments.
Pour préciser cela, Magness et son équipe se sont penchés sur l’exemple des médicaments qui traitent les maladies inflammatoires de l’intestin, en ciblant certaines cellules immunitaires qui déclenchent l’inflammation. Il s’avère que certaines cellules épithéliales expriment les mêmes gènes que ceux des cellules immunitaires ciblées par les médicaments. Il pourrait donc exister un dommage collatéral « hors cible » dans les cellules épithéliales, qui entraînerait des effets secondaires. « Beaucoup de médicaments ont de mauvais effets secondaires gastro-intestinaux, et cela pourrait être dû au fait que les médicaments affectent des cellules individuelles sur toute la longueur du tube digestif. Nous montrons où ces récepteurs sont le plus exprimés et dans quels types de cellules », a ajouté Burclaff.