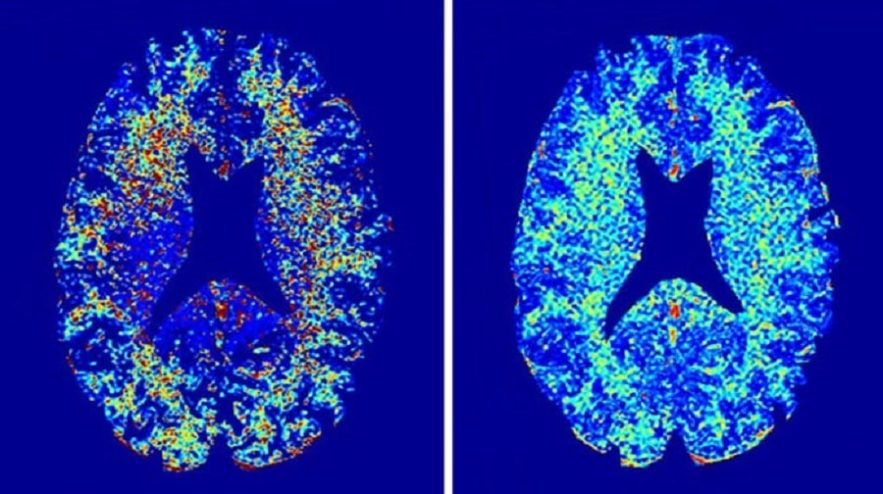
Les scanners IRM peuvent cartographier l’intérieur d’une personne avec des détails importants, mais ils ne permettent pas de connaître la composition des tissus observés. Récemment, des physiciens ont proposé une méthode pour amener l’IRM vers un nouveau domaine de sensibilité afin de détecter des biomolécules spécifiques dans les tissus, une capacité qui pourrait aider au diagnostic de la maladie d’Alzheimer et d’autres maladies.
L’avancée ne provient pas de scanners améliorés, mais de meilleures méthodes pour résoudre un problème mathématique notoirement difficile et extraire des informations déjà latentes dans les données IRM. Les nouvelles techniques, décrites ce mois-ci lors d’une réunion de l’American Physical Society, pourraient bientôt être utilisées en clinique selon Shannon Kolind, physicienne à l’Université de la Colombie-Britannique (UBC) à Vancouver.
« Je ne pense pas être trop optimiste pour dire que cela se produira dans les 5 prochaines années » dit-elle. Sean Deoni, physicien à la Brown University, affirme que « n’importe quel scanner de la planète peut le faire ».
Un IRM standard incapable de cartographier les biomolécules tissulaires
Un scanner IRM utilise des champs magnétiques et des ondes radio pour exciter les noyaux d’atomes d’hydrogène (des protons) dans des molécules d’eau, qui constituent plus de la moitié des tissus mous. Les protons agissent comme de petits aimants, et le puissant champ magnétique du scanner les fait tous pointer dans la même direction. Une impulsion d’ondes radio éloigne ensuite les protons du champ magnétique, les faisant tourner en masse comme des gyroscopes. Les protons émettent alors leurs propres ondes radio.
Le scanner suit la décroissance de ce signal dans le temps, ce qui se produit de deux manières. Les protons virevoltants se désexcitent dans la direction du champ magnétique et interagissent également les uns avec les autres. Ces deux processus sont décrits par une paire de constantes de temps, T1 et T2, qui sont comme des demi-vies dans la désintégration radioactive.
En appliquant différentes séquences d’impulsions radio, le scanner mesure les constantes de temps, qui dépendent de l’environnement chimique des molécules d’eau et, par conséquent, du type de tissu. En analysant la façon dont les constantes varient dans l’ensemble du corps, le scanner cartographie la structure des tissus mous.
Cependant, une IRM standard ne peut pas tracer de biomolécules spécifiques car elle traite chaque élément volumique de taille millimétrique dans un tissu comme s’il ne contenait qu’un seul matériau.
Pour rechercher une biomolécule particulière, les chercheurs doivent supposer que chaque voxel contient au moins deux matériaux : la biomolécule cible et tout le reste. Ils doivent ensuite mesurer au moins deux ensembles de constantes de temps : un couple pour l’eau entourant une biomolécule d’intérêt, et un autre pour l’eau plus éloignée.
Démêler ces deux ensembles est extrêmement difficile. Cela revient à écouter la même note jouée simultanément sur deux xylophones, et à dire laquelle des notes s’est estompée plus rapidement. Pour les mathématiciens, le problème est initialement compliqué, ce qui signifie qu’un peu de bruit fera varier énormément les estimations des constantes de temps qui se chevauchent.
Détecter des biomolécules spécifiques grâce aux mathématiques
En 1994, Alex MacKay, physicien à l’Université de la Colombie-Britannique, s’est attaché à séparer deux valeurs de la constante T2 pour produire la première carte cérébrale IRM montrant la myéline, la molécule adipeuse qui isole les fibres nerveuses. Au début, il a dû scanner pendant 25 minutes pour imager une seule tranche du cerveau. En 2006, Deoni a publié un protocole de pulsation plus complexe qui extrait deux valeurs pour T1 et T2 et permet de scanner tout le cerveau en 10 minutes.
Ces dernières années, Spencer et son collègue de la NIA, Mustapha Bouhrara, ont développé une approche statistique qui améliore les techniques de MacKay et de Deoni. Lors du démêlage de deux signaux décroissants, l’analyse statistique conventionnelle fournit des estimations de constantes de temps précises mais perturbées par le bruit. Spencer et Bouhrara utilisent donc une approche dite bayésienne, qui donne une distribution de probabilité pour chaque constante de temps.
L’analyse prend jusqu’à 4 heures de calcul, mais le gain pourrait être énorme. En 2017, Spencer et Bouhrara ont indiqué qu’ils pourraient cartographier des molécules appelées protéoglycanes dans le cartilage du genou, ce qui pourrait aider les médecins à déchiffrer les origines de l’arthrose.
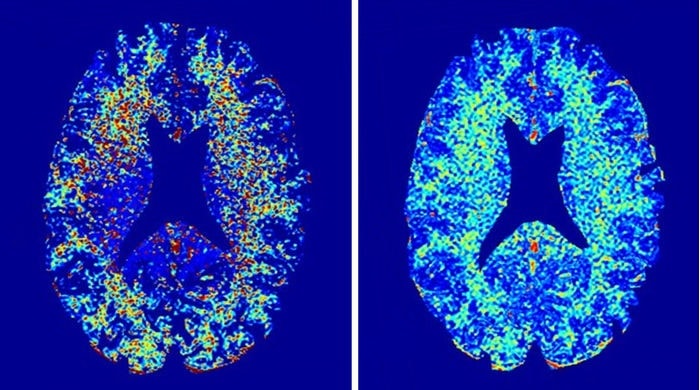


Sur le même sujet : Un nouveau scanner capable d’imager la totalité du corps humain en une seule fois a été testé avec succès
L’année dernière, ils ont rapporté que les cerveaux d’adultes atteints d’une déficience cognitive légère et de la maladie d’Alzheimer avaient moins de myéline que leurs sujets en bonne santé, ce qui montre bien que la perte de myéline pourrait jouer un rôle dans la maladie. De même, les neurologues savent depuis longtemps que les patients atteints de SEP (sclérose en plaques) développent des lésions cérébrales dans lesquelles la myéline est perdue.
En utilisant les nouveaux scanners, Kolind a découvert que certains patients atteints de SEP perdent également de la myéline ailleurs dans le cerveau, suggérant que la perte précède les lésions. Deoni et ses collègues étudient la myéline chez 1200 enfants en bonne santé au fil du temps pour voir comment elle se développe avec l’âge. Les données suggèrent que des facteurs sociaux tels que la richesse peuvent également entraîner des différences de myélinisation explique Deoni.
Spencer dit que son équipe est impatiente de passer aux applications de diagnostic. « Notre espoir a toujours été de transformer cela en un outil clinique plutôt qu’en un simple jouet de recherche ». À cette fin, Kolind et d’autres membres de l’Université de Colombie-Britannique examinent des sujets en bonne santé pour élaborer une carte de la myéline normale, contre laquelle des balayages anormaux pourraient apparaître.
Néanmoins, les chercheurs doivent comparer les méthodes avec les données cliniques et les échantillons de tissus pour des maladies spécifiques, ce qui pourrait prendre des années.