Après s’être imposé en véritable maître face aux champions du monde d’échecs, de Go et de shogi avec son intelligence artificielle, l’entreprise DeepMind de Google montre de nouveau ses talents avec AlphaFold, une IA capable de prédire la structure 3D des protéines à partir d’une séquence d’acides aminés donnée en entrée. Lors du 13ème Critical Assessment of Structure Prediction (CASP), une compétition bisannuelle sur la prédiction de la conformation 3D protéique, AlphaFold a montré une précision supérieure à celle des autres concurrents.
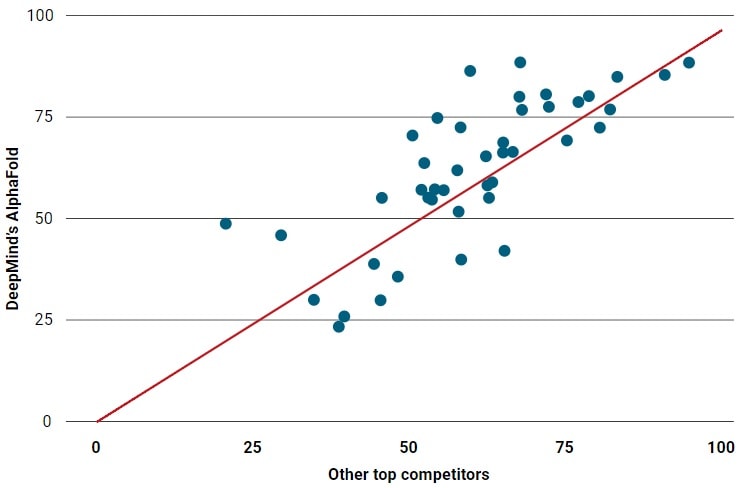
Les équipes concurrentes ont reçu la séquence linéaire d’acides aminés correspondant à 90 protéines pour lesquelles la forme 3D est connue, mais non encore publiée. Les équipes ont ensuite calculé le déroulement de ces séquences. Bien que DeepMind, basé à Londres, n’ait jamais rejoint cette compétition par le passé, les prévisions de son logiciel AlphaFold étaient en moyenne plus précises que celles de ses 97 concurrents.
À la fin de l’épreuve, il n’a pas été difficile de départager les différents concurrents. Pour les séquences protéiques pour lesquelles aucune autre information n’était connue — 43 sur 90 — AlphaFold a effectué la prédiction la plus précise 25 fois. Cela a dépassé de loin le candidat en deuxième position, qui n’a remporté que 3 des 43 tests.
AlphaFold aurait donc écrasé la concurrence ? Eh bien, pas exactement. Lorsque que l’on regarde le nombre de gains ou de pertes d’AlphaFold dans chaque cas, les résultats semblent beaucoup plus proches. AlphaFold a remporté de nombreuses manches, avec une marge moyenne d’amélioration de la précision de 15% par rapport aux autres groupes lors des 43 tests les plus difficiles, a déclaré John Moult, principal organisateur de la CASP et biologiste en informatique à l’Université du Maryland.
David Baker, organisateur, participant et expert en modélisation informatique du CASP à l’Université de Washington à Seattle, note que les scientifiques de DeepMind s’appuient sur deux stratégies d’algorithme mises au point par d’autres. Premièrement, en comparant de vastes données génomiques sur d’autres protéines, AlphaFold a été en mesure de mieux déchiffrer les paires d’acides aminés les plus susceptibles de se retrouver proches les unes des autres dans des protéines repliées.
Sur le même sujet : Google a ajouté la capacité d’imagination à l’intelligence artificielle DeepMind
Deuxièmement, des comparaisons connexes les ont également aidés à évaluer la distance la plus probable entre des paires d’acides aminés voisins et les angles auxquels ils se sont liés à leurs voisins. Les deux approches donnent de meilleurs résultats avec le nombre de données qu’elles évaluent, ce qui les rend plus aptes à tirer parti d’algorithmes d’apprentissage automatique, tels que AlphaFold, qui résolvent les problèmes en traitant des ensembles de données volumineux.
Les scientifiques de DeepMind « sont extrêmement doués pour l’apprentissage automatique et forment une équipe superbe, avec plus de ressources que la plupart des autres groupes académiques » explique Baker. « Donnez-leur du crédit. Après tout, ils sont arrivés de nulle part » conclut John Moult, un autre organisateur du PCSA et biologiste en informatique à l’Université du Maryland.