À la fin du XIVème siècle, l’Europe subit l’un des plus grands cataclysmes de son histoire : la seconde pandémie de peste noire. Appelée la Mort Noire, cette pandémie va décimer plus de la moitié de la population européenne. Pour mieux comprendre l’arrivée et l’évolution de la bactérie responsable de la peste, Yersinia pestis, les biologistes étudient le génome de ses différentes souches bactériennes. Et récemment, une équipe de chercheurs a pu analyser d’anciens génomes bactériens issus de restes humains datant de l’épidémie afin de reconstituer le parcours de Y. pestis en Europe.
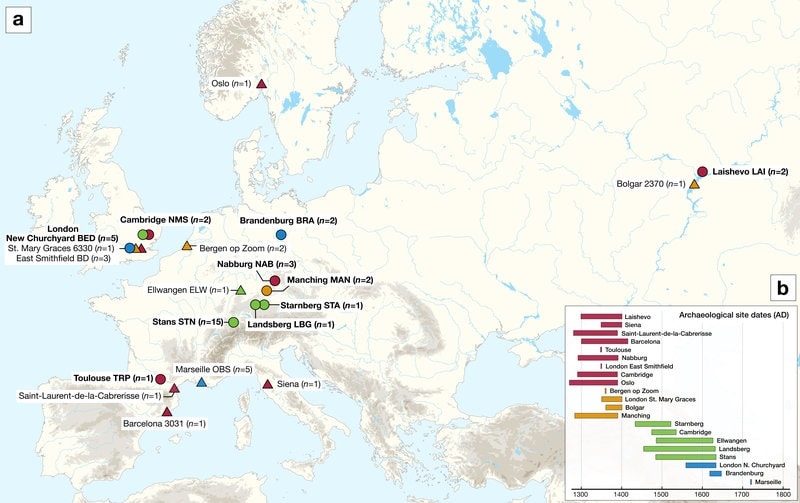
Une équipe internationale de chercheurs a analysé les restes de dix sites archéologiques situés en Angleterre, en France, en Allemagne, en Russie et en Suisse, afin de mieux comprendre les différents stades de la deuxième pandémie de peste (XIVe-XVIIIe siècles) et la diversité génétique de Yersinia pestis durant et après la mort noire.
Dans une étude publiée dans Nature Communications, les chercheurs ont reconstitué 34 génomes de Y. pestis, en retraçant l’historique génétique de la bactérie, qui ont permis de mieux comprendre l’initiation et la progression de la deuxième pandémie de peste en Europe.
La deuxième pandémie de peste, qui a commencé avec la peste noire au milieu du XIVe siècle et s’est poursuivie avec des épidémies dévastatrices en Europe et dans les environs jusqu’au XVIIIe siècle, a décimé le continent, causant la mort de près de 60% de la population. Mais d’où vient cette souche de Yersinia pestis, la bactérie responsable de la peste ? Et comment a-t-elle évolué et s’est développée une fois arrivée en Europe ?
Un point d’entrée unique de Yersinia pestis en Europe
En dépit de l’omniprésence de la peste noire dans les textes historiques et de l’imagination populaire, le point d’entrée de la bactérie Y. pestis à cette époque et son itinéraire emprunté en Europe restent flous, en raison du manque de données sur les premiers foyers et d’une rareté générale des données concernant les premiers génomes de la bactérie.
Dans la présente étude, les chercheurs ont reconstitué les génomes de la peste à partir des dents de 34 personnes, dont deux de Laishevo, dans la région de la Volga en Russie, et ont découvert une seule souche ancestrale de laquelle proviennent toutes les souches de la deuxième pandémie. De plus, les chercheurs ont observé une absence de diversité génomique dans les échantillons prélevés pendant la peste noire.

« Ces résultats indiquent une entrée unique de Y. pestis en Europe par l’Est » explique le premier auteur, Maria Spyrou. « Cependant, il est possible que des interprétations supplémentaires soient révélées lors de futures découvertes d’une diversité non échantillonnée en Eurasie occidentale ».
La persistance de la peste noire en Europe grâce à plusieurs souches bactériennes
Bien que les chercheurs aient constaté que la peste noire à l’échelle européenne fut probablement causée par une seule souche, l’analyse des génomes de la partie tardive de la pandémie montre l’émergence d’une lignée présentant une diversité génétique plus élevée.
« Au cours de la phase ultérieure de la deuxième pandémie, nous assistons au développement de plusieurs branches en Europe, ce qui suggère que la peste a été maintenue dans différents foyers locaux » déclare Marcel Keller. « Aucun descendant moderne de cette lignée n’a été trouvé à ce jour, ce qui pourrait indiquer l’extinction de ces réservoirs ».

Sur le même sujet : Une épidémie mystérieuse datant du 16ème siècle, qui a presque anéanti le Mexique, a finalement été identifiée
Les chercheurs ont également identifié une délétion comprenant deux gènes liés à la virulence dans les génomes de cette deuxième lignée. Fait intéressant, les génomes des derniers stades de la première pandémie de peste ont montré une délétion dans la même région.
« Étant donné que cette suppression s’est produite dans les lignées de la première et de la deuxième pandémie, les deux maintenant éteintes, déterminer comment l’impact de ces gènes sur la persistance chez l’Homme et les puces constituerait un domaine important pour une étude future » commente Kirsten Bos, responsable du groupe de recherche du Max Planck Institut pour la science de l’histoire humaine.
Vers une meilleure compréhension de la seconde pandémie de peste
L’étude offre de nouvelles perspectives sur l’initiation et la progression de la deuxième pandémie de peste et s’ajoute de manière significative à la base de données des génomes de Y. pestis déjà publiés. « Nous avons montré qu’une analyse approfondie des génomes anciens de Y. pestis peut fournir des informations uniques sur la micro-évolution d’un agent pathogène sur une période de plusieurs centaines d’années » explique l’auteur principal Johannes Krause.
À l’avenir, l’intégration de ces données dans les efforts de modélisation de la maladie, conjointement avec des données provenant d’autres domaines tels que la climatologie, l’épidémiologie et l’histoire, sera importante pour mieux comprendre la deuxième pandémie de peste.


