Tout comme l’étude de l’hydrogène — l’atome le plus simple du tableau périodique des éléments — a permis de mieux comprendre les atomes plus complexes, l’étude d’une cellule vivante parmi les plus simples devrait révéler des mécanismes applicables à des systèmes vivants plus complexes. Sur la base de ce raisonnement, une équipe de chercheurs américains a entrepris de créer un modèle informatique d’une « cellule minimale », dépourvue de tous les gènes non essentiels, afin d’étudier son comportement et ainsi mettre en évidence les mécanismes fondamentaux qui sous-tendent la vie.
La cellule est l’unité fondamentale de la vie, une unité partagée par les plantes, les animaux et les bactéries. Comprendre le fonctionnement d’une cellule dans son intégralité est indispensable pour la recherche biomédicale. Une description complète de l’état d’une cellule nécessite toutefois de connaître sa taille, sa forme, ses composants, ses réactions intra- et extracellulaires, le tout en fonction du temps et de sa croissance. Or, la fonction d’un grand nombre de gènes reste encore inconnue aujourd’hui, ce qui rend la simulation complète de l’état d’une cellule particulièrement difficile.
Les modèles les plus complets qui ont été développés jusqu’à présent étaient basés sur les bactéries Mycoplasma genitalium et Escherichia coli. M. genitalium présente le plus petit génome connu : il ne compte que 525 gènes (pour comparaison, une cellule humaine en comporte plus de 20 000 !). À partir de ce génome restreint, des chercheurs ont réussi à développer en 2019 une cellule bactérienne, Syn3A, composée de seulement 493 gènes sur un seul chromosome circulaire, dont 452 gènes codaient pour des protéines. Une équipe a cette fois entrepris de modéliser le fonctionnement de cette cellule minimale sur un cycle de vie complet, incluant notamment sa division. Leurs résultats ont été publiés dans la revue Cell.
Une fenêtre ouverte sur le fonctionnement cellulaire
L’objectif de cette recherche était de mettre au point un système permettant de prédire comment les modifications du génome, des conditions de vie ou des caractéristiques physiques des cellules vivantes influent sur leur fonctionnement. « Ce qui est nouveau ici, c’est que nous avons développé un modèle cinétique tridimensionnel entièrement dynamique d’une cellule minimale vivante qui imite ce qui se passe dans la cellule réelle », a déclaré Zaida Luthey-Schulten, professeure de chimie à l’Université de l’Illinois, codirectrice du Center for the Physics of Living Cells de la National Science Foundation et co-auteure de l’étude.
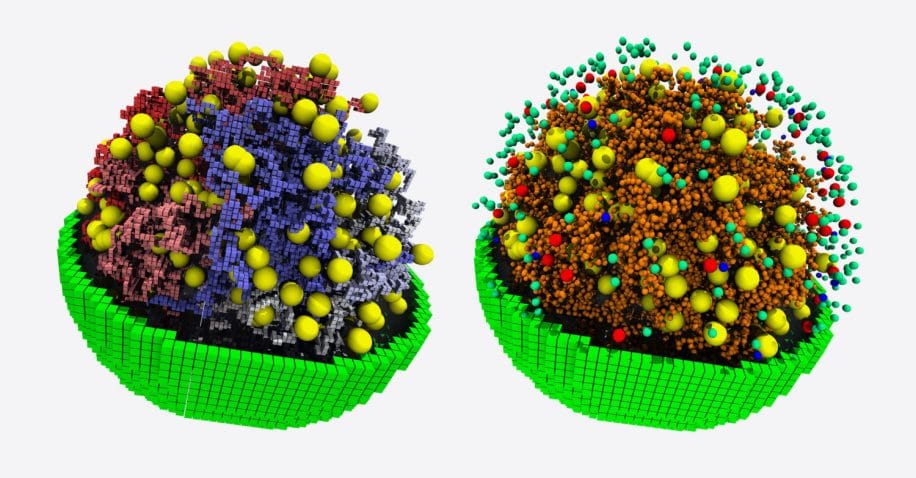
La cellule que Luthey-Schulten et ses collègues ont modélisée, baptisée JCVI-syn3A, repose sur un génome simplifié au maximum, ne comportant que les gènes nécessaires pour répliquer son ADN, croître, se diviser et exécuter la plupart des autres fonctions qui définissent la vie. Ils sont ainsi parvenus à cartographier très précisément l’emplacement et les caractéristiques chimiques de milliers de composants cellulaires à l’échelle atomique.
À partir de ce modèle, les scientifiques peuvent suivre en détail toutes les réactions chimiques qui se produisent au sein de la cellule et observer comment tous les composants interagissent et changent en réponse à des signaux internes et externes ; les chercheurs disposent désormais d’une fenêtre ouverte directement sur le fonctionnement interne d’une cellule (les molécules qui entrent en jeu, la quantité d’énergie nécessaire à chaque processus, le temps de réaction, etc.).
Pour parvenir à un tel modèle, ils ont dû intégrer de nombreux paramètres : les caractéristiques physiques et chimiques de l’ADN de la cellule, ses lipides, ses acides aminés, ainsi que toute la machinerie de transcription, de traduction et de fabrication des protéines. Ils ont également dû modéliser la diffusion de chaque composant à travers la cellule (nutriments, produits, déchets et autres molécules), de manière à évaluer l’énergie nécessaire à chaque étape du cycle cellulaire. À partir des simulations, l’équipe a pu faire des découvertes surprenantes sur la physiologie et le cycle de reproduction de cette cellule.
Une régulation optimale des apports énergétiques
« Nous avons simulé toutes les réactions chimiques à l’intérieur d’une cellule minimale, de sa naissance jusqu’au moment où elle se divise deux heures plus tard », a précisé Zane Thornburg, co-auteur de l’étude. Leur modèle a notamment révélé que la cellule utilisait la majeure partie de son énergie pour importer des ions et des molécules essentiels à travers sa membrane cellulaire. Ce résultat est peu surprenant étant donné que les mycoplasmes — les bactéries dont dérive le modèle — tirent la plupart des éléments essentiels à leur survie d’autres organismes. Les déséquilibres issus de demandes énergétiques accrues étaient quant à eux immédiatement compensés par un ralentissement d’autres processus.
Les simulations ont également permis de calculer la durée de vie naturelle des ARN messagers, sur lesquels repose la synthèse des protéines. Elles ont également révélé une relation entre le taux de synthèse des lipides et des protéines membranaires et les modifications de la surface membranaire et du volume cellulaire. L’équipe a également noté que pour se diviser aussi rapidement qu’elle le fait, la cellule a besoin d’une enzyme appelée transaldolase ; cependant, ils n’ont décelé aucune trace de sa présence, ce qui suggère que la cellule a peut-être développé une voie métabolique alternative rendant cette enzyme inutile. Une autre possibilité est que cette enzyme existe, mais qu’elle ne ressemble pas à une transaldolase ordinaire, a précisé à Quanta Magazine John Glass, co-auteur de l’étude.
Ce modèle cellulaire ouvre ainsi la voie à de très nombreuses expériences, visant à mieux comprendre le métabolisme cellulaire et à l’améliorer. En injectant des molécules supplémentaires ou en changeant les conditions environnementales de la cellule, les chercheurs pourront déterminer plus finement les éléments qui sont essentiels à sa survie. Ils prévoient également d’améliorer leur modèle pour simuler plusieurs cycles cellulaires d’affilée. « Ce modèle et d’autres modèles plus sophistiqués à venir nous aideront à mieux comprendre les principes fondamentaux de la vie », a déclaré Luthey-Schulten.