En 2015, la production mondiale de plastiques s’élevait à 322 millions de tonnes. Aujourd’hui, plus de 45% du plastique fabriqué est utilisé à des fins éphémères, tandis que les déchets plastiques représentent une part importante de la pollution environnementale. De nombreux processus sont actuellement à l’étude pour optimiser le recyclage et la transformation écologique du polymère. Récemment, la mise au point accidentelle d’une enzyme dégradant le plastique pourrait ouvrir une voie prometteuse aux efforts déployés en ce sens.
Très peu biodégradables, les déchets plastiques, toujours plus nombreux, conduisent à une importante pollution planétaire impactant autant les écosystèmes marins que terrestres. En 2016, en analysant des échantillons de sol d’une usine de recyclage de plastique, une équipe de biologistes japonais de l’université de Kyoto a découvert l’existence d’une bactérie nommée « Ideonella sakaiensis ». La particularité de cette dernière réside dans le fait qu’elle ait évolué afin de pouvoir dégrader et utiliser le plastique PET (téréphtalate d’éthylène) comme source de carbone et d’énergie.
En étudiant Ideonella sakaiensis et ses mécanismes cellulaires, les scientifiques ont accidentellement synthétisé une nouvelle enzyme bien plus performante que celle utilisée par la bactérie et qui pourrait offrir une véritable solution à la déferlante plastique actuelle. « La sérendipité joue souvent un rôle important dans la recherche scientifique fondamentale et notre découverte n’y fait pas exception » explique John McGreehan, biologiste structural à l’université de Portsmouth (Royaume-Uni). « Cette découverte non anticipée suggère qu’il est possible d’améliorer davantage ces enzymes, nous rapprochant d’une solution de recyclage pour chaque montagne de déchets plastiques ».
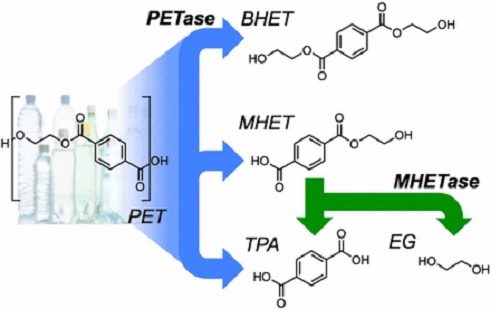
L’équipe de McGreehan, incluant des chercheurs de l’US Department of Energy’s National Renewable Energy Laboratory (NREL), ont synthétisé la nouvelle enzyme en étudiant la structure cristalline de la PETase – l’enzyme permettant à Ideonella sakaiensis de dissoudre le plastique PET. Les résultats ont été publiés dans la revue Proceedings of the National Academy of Sciences (PMNAS).
La structure complexe du PET, obtenue par polycondensation de l’acide téréphtalique avec l’éthylène glycol, le rend extrêmement peu biodégradable et nécessite plusieurs siècles pour une dissolution partielle. Là où la PETase de Ideonella sakaiensis permet une dégradation en quelques semaines maximum, la nouvelle PETase améliorée ramène ce délai à quelques jours. « Après seulement 96h, nous pouvons clairement voir au microscope électronique que la PETase dégrade le PET ; et nous la testons avec de véritables déchets plastiques, tels que ceux trouvés dans les océans ou les décharges » indique le biologiste Bryon Donohoe (NREL).
Afin d’étudier l’efficacité de la PETase au niveau moléculaire, les auteurs ont utilisé la diffraction par rayons X pour générer un modèle 3D de l’enzyme en ultra-haute résolution. Cette analyse a permis aux chercheurs d’identifier les sites enzymatiques actifs impliqués dans la fixation et la dégradation du PET. En obtenant une image claire du mécanisme d’action de la PETase, les scientifiques ont ainsi pu déterminer des façons de l’améliorer. « Être capable d’observer le fonctionnement interne de cette catalyse biologique nous a donné les plans pour fabriquer une enzyme plus rapide et plus efficace » explique McGreehan.
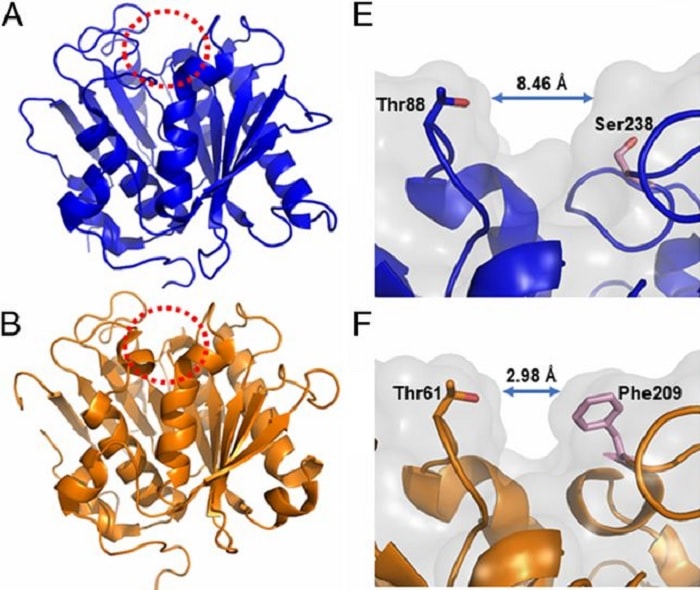
Les auteurs ont tenté de modifier la PETase pour la rapprocher du mécanisme enzymatique de la cutinase – une enzyme dégradant la cutine, un biopolymère lipidique présent chez les plantes et dont la structure ressemble à celle du PET. En raccourcissant les liaisons entre les sites actifs de la PETase, ils se sont aperçus que la catalyse du plastique obtenu était bien plus performante que prévu.
« De façon surprenante, nous avons découvert que la PETase mutante surpassait la PETase naturelle pour dégrader le PET » explique Nic Rorrer, chimiste des polymères (NREL). « Comprendre comment le PET se fixe aux sites catalytiques de la PETase grâce à des outils informatiques, nous a aidé à identifier les raisons de cette performance améliorée. Considérant ces résultats, il est certain que son potentiel pourra encore être augmenté ».
Bien que la PETase mutante soit plus efficace de 20% que la PETase initiale, les chercheurs expliquent que la découverte est importante, dans le sens où nous savons maintenant concrètement comment en améliorer les performances. Cela signifie que, dans le futur, ces enzymes pourront être optimisées de manière à dégrader le PET et d’autres matériaux bien plus rapidement. En effet, la PETase mutante s’est également révélée apte à dégrader un dérivé du PET : le PEF (polyethylene-2,5-furandicarboxylate).
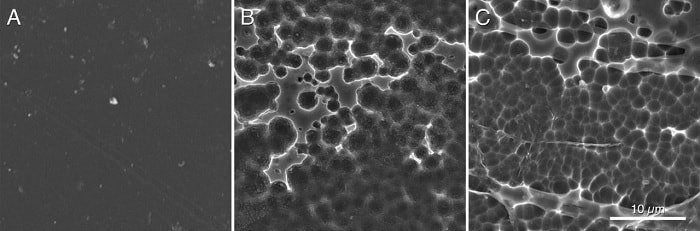
Un important travail attend encore les scientifiques avant que cette nouvelle enzyme puisse être produite en masse et permette véritablement d’offrir une méthode de recyclage à grande échelle. Cependant, maintenant que l’on connait son fonctionnement, son utilisation n’est plus qu’une question de temps. « Nous savons que cette PETase n’est pas encore totalement optimisée pour dégrader le PET. Et donc maintenant, il est temps d’utiliser les outils de l’ingénierie protéique pour poursuivre son amélioration » conclut Gregg Beckman (NREL).


