Depuis l’apparition du coronavirus SARS-CoV-2, les virologues l’étudient sous toutes les coutures afin de comprendre en détail sa structure, sa dynamique et ses mécanismes d’infection cellulaire. Plus d’un an après le début de la pandémie, les chercheurs sont enfin capables de dresser un tableau complet des stratégies virales du SARS-CoV-2, bien que des zones d’ombre persistent encore.
Depuis le début de la pandémie de COVID-19, les scientifiques ont développé une compréhension détaillée de la façon dont le SARS-CoV-2 infecte les cellules. En séparant le processus d’infection, ils espèrent trouver de meilleurs moyens de l’interrompre grâce à des traitements et des vaccins améliorés, et apprendre pourquoi les dernières souches, telles que la variante Delta, sont plus transmissibles. Ce qui a émergé de 19 mois de travail, soutenu par des décennies de recherche sur les coronavirus, est un compte rendu détaillé de la façon dont le SARS-CoV-2 envahit les cellules humaines.
Les chercheurs ont découvert des adaptations clés qui aident le virus à s’accrocher aux cellules humaines avec une force surprenante, puis à se cacher une fois à l’intérieur. Plus tard, lorsqu’il quitte les cellules, le SARS-CoV-2 exécute une étape de traitement cruciale pour préparer ses particules à infecter encore plus de cellules humaines. Ce sont quelques-uns des outils qui ont permis au virus de se propager si rapidement et de faire des millions de morts.
Le rôle essentiel des glycanes
Le coronavirus arbore un dense manteau de sucre. De nombreux virus ont des glycanes recouvrant leurs protéines externes, les camouflant auprès du système immunitaire humain. Mais l’année dernière, le groupe du laboratoire de Rommie Amaro — chimiste biophysicien informaticien à l’université de Californie à San Diego — et ses collaborateurs ont créé la visualisation la plus détaillée à ce jour de cette couche, basée sur des données structurelles et génétiques et rendue atome par atome par un superordinateur. Le 22 mars 2020, elle a publié la simulation sur Twitter. En moins d’une heure, un chercheur demandait dans un commentaire : quelle était la boucle nue et sans manteau qui dépassait du sommet de la protéine ?
Amaro n’en avait aucune idée. Mais dix minutes plus tard, le biologiste structural Jason McLellan de l’Université du Texas à Austin intervint : la boucle non couverte était un domaine de liaison au récepteur (RBD), l’une des trois sections de la protéine de pointe qui se lient aux récepteurs des cellules humaines. Dans la simulation d’Amaro, lorsque le RBD s’est élevé au-dessus du manteau de glycane, deux glycanes sont apparus pour le verrouiller. Quand Amaro a muté les glycanes dans le modèle informatique, le RBD s’est effondré.
L’équipe de McLellan a mis au point un moyen d’essayer la même expérience en laboratoire et, en juin 2020, les collaborateurs avaient signalé que la mutation des deux glycanes réduisait la capacité de la protéine de pointe à se lier à un récepteur cellulaire humain – un rôle que personne n’avait auparavant reconnu dans les coronavirus, déclare McLellan. Il est possible que l’extraction de ces deux sucres réduise l’infectiosité du virus, selon Amaro, bien que les chercheurs n’aient pas encore le moyen de le faire.
La protéine de pointe : l’arme d’abordage du SARS-CoV-2
Cela commence par les pointes. Chaque virion (particule virale) du SARS-CoV-2 a une surface externe parsemée de 24 à 40 protéines de pointe disposées au hasard qui sont la clé de la fusion avec les cellules humaines. Pour d’autres types de virus, comme la grippe, les protéines de fusion externes sont relativement rigides. Les pointes du SARS-CoV-2, cependant, sont extrêmement flexibles et s’articulent en trois points, selon les travaux publiés en août 2020 par le biochimiste Martin Beck de l’Institut Max Planck de biophysique de Francfort, en Allemagne, et ses collègues.
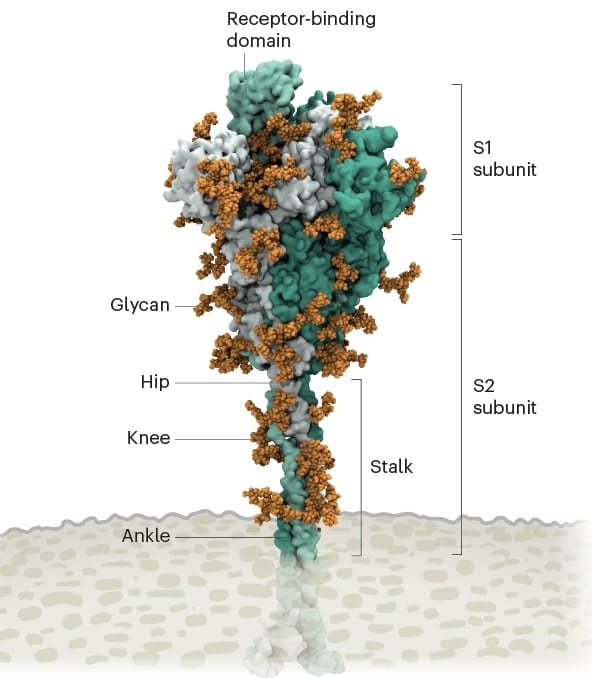
Cela permet aux pointes de basculer, de se balancer et de tourner, ce qui pourrait leur permettre de scanner plus facilement la surface cellulaire et de permettre à plusieurs pointes de se lier à une cellule humaine. Il n’y a pas de données expérimentales similaires pour d’autres coronavirus, mais comme les séquences de protéines de pointe sont hautement conservées au cours de l’évolution, il est juste de supposer que le trait est partagé.
Au début de la pandémie, les chercheurs ont confirmé que les RBD des protéines de pointe du SARS-CoV-2 se fixent à une protéine familière appelée récepteur ACE2, qui orne l’extérieur de la plupart des cellules de la gorge et des poumons humains. Ce récepteur est également le point d’ancrage du SARS-CoV, le virus qui cause le syndrome respiratoire aigu sévère (SRAS). Mais par rapport au SARS-CoV, le SARS-CoV-2 se lie à l’ACE2 environ 2 à 4 fois plus fortement, car plusieurs changements dans le RBD stabilisent ses points chauds de liaison aux virus.
Des variantes inquiétantes du SARS-CoV-2 ont tendance à avoir des mutations dans la sous-unité S1 de la protéine de pointe, qui héberge les RBD et est responsable de la liaison au récepteur ACE2 (une deuxième sous-unité de pointe, S2, provoque la fusion virale avec la membrane de la cellule hôte). La variante Alpha, par exemple, comprend dix changements dans la séquence des protéines de pointe, ce qui fait que les RBD sont plus susceptibles de rester en position « haut ». La variante Delta, qui se répand maintenant dans le monde, héberge de multiples mutations dans la sous-unité S1, dont trois dans le RBD qui semblent améliorer la capacité du RBD à se lier à l’ACE2 et à échapper au système immunitaire.
Une entrée cellulaire restreinte
Une fois que les pointes virales se lient à l’ACE2, d’autres protéines à la surface de la cellule hôte initient un processus qui conduit à la fusion des membranes virales et cellulaires. Le virus qui cause le SRAS, le SARS-CoV, utilise l’une des deux enzymes protéases de l’hôte pour s’introduire : TMPRSS2 (prononcé « température deux ») ou la cathepsine L. TMPRSS2 est la voie la plus rapide, mais le SARS-CoV entre souvent à la place par un endosome — une bulle entourée de lipides — qui repose sur la cathepsine L. Cependant, lorsque les virions pénètrent dans les cellules par cette voie, des protéines antivirales peuvent les piéger.
Le SARS-CoV-2 diffère du SARS-CoV, car il utilise efficacement TMPRSS2, une enzyme trouvée en grande quantité à l’extérieur des cellules respiratoires. Tout d’abord, TMPRSS2 coupe un site sur la sous-unité S2 de la pointe. Cette coupe expose une série d’acides aminés hydrophobes qui s’enfouissent rapidement dans la membrane la plus proche, celle de la cellule hôte. Ensuite, la pointe étendue se replie sur elle-même, comme une fermeture éclair, forçant les membranes virales et cellulaires à fusionner. Le virus éjecte alors son génome directement dans la cellule.
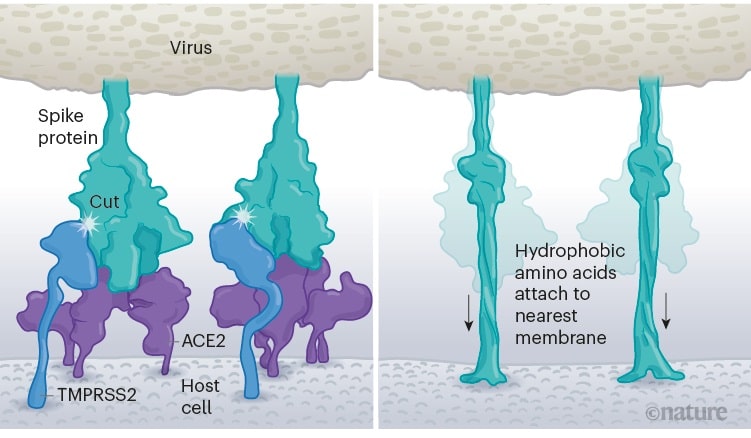
En envahissant de cette manière, le SARS-CoV-2 infecte plus rapidement que le SARS-CoV et évite d’être piégé dans les endosomes, selon des travaux publiés en avril par Barclay et ses collègues de l’Imperial College de Londres. L’entrée rapide du virus à l’aide de TMPRSS2 explique pourquoi la chloroquine, un médicament contre le paludisme, n’a pas fonctionné dans les essais cliniques en tant que traitement COVID-19, malgré les premières études prometteuses en laboratoire. En effet, la chloroquine est un perturbateur endosomal, et n’a donc pas d’effets dans ce cas-ci.
La découverte indique également que les inhibiteurs de protéase constituent une option thérapeutique prometteuse pour empêcher un virus d’utiliser TMPRSS2, la cathepsine L ou d’autres protéases pour pénétrer dans les cellules hôtes. Un inhibiteur de TMPRSS2, le camostat mésylate, qui est approuvé au Japon pour traiter la pancréatite, a bloqué l’entrée du virus dans les cellules pulmonaires, mais le médicament n’a pas amélioré les résultats des patients lors d’un essai clinique initial.
Éliminer la concurrence cellulaire
Une fois que le virus a injecté son génome d’ARN dans la cellule, les ribosomes du cytoplasme traduisent deux sections d’ARN viral en longues chaînes d’acides aminés, qui sont ensuite découpées en 16 protéines, dont beaucoup sont impliquées dans la synthèse de l’ARN. Plus tard, davantage d’ARN sont générés qui codent pour un total de 26 protéines virales connues, y compris les protéines structurelles utilisées pour fabriquer de nouvelles particules virales, telles que la pointe, et d’autres protéines accessoires. De cette façon, le virus commence à produire des copies de son propre ARN messager. Mais il a besoin de la machinerie de la cellule pour traduire ces ARNm en protéines.
Les coronavirus prennent le contrôle de cette machine de plusieurs manières. La virologue Noam Stern-Ginossar et son équipe de l’Institut des sciences Weizmann à Rehovot, en Israël, ont zoomé sur trois mécanismes par lesquels le SARS-CoV-2 supprime la traduction de l’ARNm de l’hôte en faveur du sien. Aucun n’est exclusif à ce virus, mais la combinaison, la vitesse et l’ampleur des effets semblent uniques, explique Stern-Ginossar.
Premièrement, le virus élimine la compétition : la protéine virale Nsp1, l’une des premières protéines traduites à l’arrivée du virus, recrute des protéines hôtes pour hacher systématiquement tous les ARNm cellulaires qui n’ont pas d’étiquette virale. Lorsque l’équipe de Stern-Ginossar a mis cette même étiquette à l’extrémité d’un ARNm hôte, l’ARNm n’a pas été découpé. Deuxièmement, l’infection réduit la traduction globale des protéines dans la cellule de 70%. Nsp1 est à nouveau le principal coupable, bloquant cette fois physiquement le canal d’entrée des ribosomes afin que l’ARNm ne puisse pas pénétrer à l’intérieur, selon les travaux de deux équipes de recherche. La petite capacité de traduction qui reste est dédiée aux ARN viraux, explique Stern-Ginossar.
Enfin, le virus arrête le système d’alarme de la cellule. Cela se produit de nombreuses manières, mais l’équipe de Stern-Ginossar a identifié un mécanisme clair pour le SARS-CoV-2 : le virus empêche l’ARNm cellulaire de sortir du noyau, y compris des instructions pour les protéines destinées à alerter le système immunitaire de l’infection. Une deuxième équipe a confirmé ce résultat, et a de nouveau pointé du doigt Nsp1 : la protéine semble bloquer les canaux de sortie dans le noyau afin que rien ne puisse s’échapper.
Parce que les transcrits génétiques ne peuvent pas sortir du noyau, les cellules infectées ne libèrent pas beaucoup d’interférons — ce sont des protéines de signalisation qui alertent le système immunitaire de la présence d’un virus. Le SARS-CoV-2 est particulièrement efficace pour arrêter ce système d’alarme : par rapport à d’autres virus respiratoires, dont le SARS-CoV et le virus respiratoire syncytial, l’infection par le SARS-CoV-2 induit des niveaux d’interférons significativement plus faibles. Et en juin dernier, des chercheurs ont signalé des mutations dans la variante Alpha qui semblent lui permettre de maîtriser encore plus efficacement la production d’interféron.
Au moment où le système immunitaire réalise qu’il y a un virus, il y en a tellement que les protéines de réponse immunitaire inondent parfois la circulation sanguine à un rythme plus rapide que la normale, ce qui peut causer des dommages. Les médecins ont constaté au début de la pandémie que certaines personnes atteintes de COVID-19 qui tombent très malades sont lésées par une réponse immunitaire hyperactive au SARS-CoV-2, ainsi que par le virus lui-même. Certains traitements éprouvés agissent en atténuant cette réponse immunitaire.
Un remodelage cellulaire complet
Une fois que le virus a pris en charge la traduction de l’hôte, il commence plusieurs changements, remodelant considérablement l’intérieur et l’extérieur de la cellule selon ses besoins. Premièrement, certaines des protéines de pointe virales nouvellement fabriquées se déplacent à la surface de la cellule et sortent de la membrane de la cellule hôte. Là, ils activent un canal ionique calcium hôte, qui expulse un revêtement gras à l’extérieur de la cellule — le même revêtement que l’on trouve sur les cellules qui fusionnent naturellement, telles que les cellules musculaires.
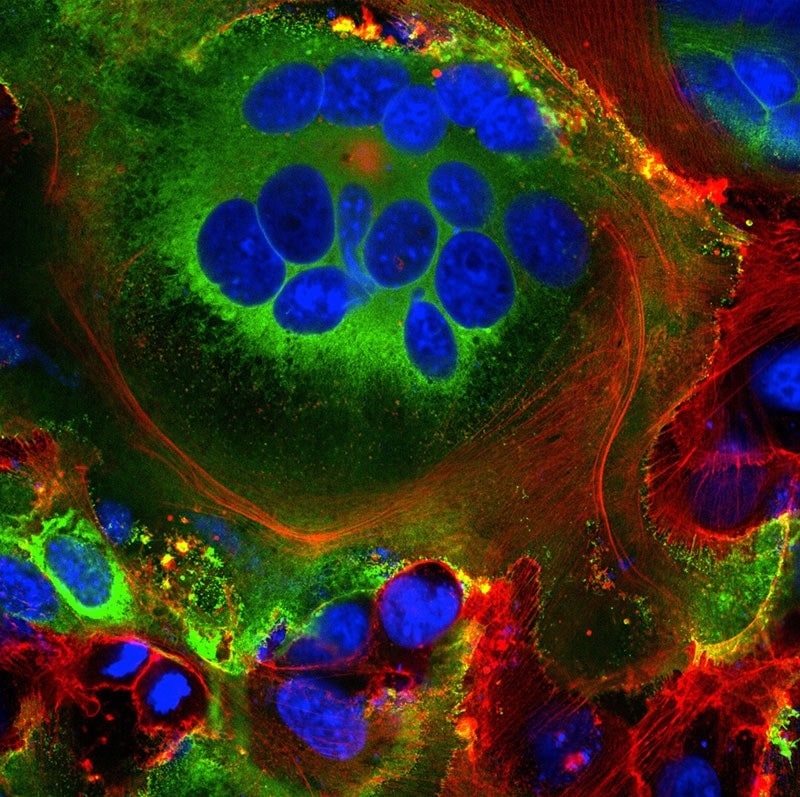
À ce stade, la cellule infectée fusionne avec les cellules voisines exprimant l’ACE2, se développant en cellules respiratoires individuelles massives contenant jusqu’à 20 noyaux. Ces structures fusionnées, appelées syncytia, sont induites par des infections virales telles que le VIH et le virus de l’herpès simplex, mais pas par le virus du SRAS, explique le biologiste moléculaire Mauro Giacca du King’s College de Londres, qui a dirigé l’équipe qui a publié les résultats en avril. Il émet l’hypothèse que la formation de syncytia permet aux cellules infectées de se développer pendant de longues périodes, produisant de plus en plus de virions.
Une deuxième équipe, dirigée par le chercheur Qiang Sun de l’Académie chinoise des sciences médicales à Pékin, a découvert que certaines cellules infectées par la COVID-19 forment même des syncytia avec des lymphocytes — l’une des propres cellules immunitaires du corps. Il s’agit d’un mécanisme connu d’évasion immunitaire par les cellules tumorales, mais pas par les virus. Cela suggère que les cellules infectées évitent la détection immunitaire en s’accrochant simplement et en fusionnant avec les éclaireurs immunitaires à proximité.
À l’intérieur de la cellule, encore plus de changements se produisent. Comme d’autres coronavirus, le SARS-CoV-2 transforme le long et mince réticulum endoplasmique (RE), un réseau de membranes plates impliquées dans la synthèse et le transport des protéines, en sphères à double membrane, comme si le RE soufflait des bulles. Ces vésicules à double membrane (DMV) pourraient fournir un endroit sûr pour la réplication et la traduction de l’ARN viral, le protégeant des capteurs immunitaires innés dans la cellule, mais cette hypothèse est toujours à l’étude.
Les protéines impliquées dans la fabrication des DMV pourraient être de bonnes cibles médicamenteuses, car elles semblent être nécessaires à la réplication virale. Par exemple, une protéine hôte, TMEM41B, est nécessaire pour mobiliser le cholestérol et d’autres lipides afin d’élargir les membranes du RE, pour que toutes les parties du virus s’y adaptent. La protéine transmembranaire du coronavirus Nsp3 pourrait également être une cible : elle crée un pore en forme de couronne dans les parois des DMV pour faire sortir l’ARN viral nouvellement fabriqué.
La plupart des virus qui ont une enveloppe externe forment cette caractéristique en s’assemblant directement au bord de la cellule, cooptant une partie de la propre membrane plasmique de la cellule lors de sa sortie. Mais les protéines de coronavirus nouvellement fabriquées empruntent un chemin différent. Pendant des années, des preuves ont suggéré que les coronavirus sont transportés hors de la cellule par le complexe de Golgi, un organite qui fonctionne comme un bureau de poste, emballant des molécules dans des membranes et les envoyant vers d’autres parties de la cellule.
Là, le virus forme une enveloppe lipidique à partir de la membrane du complexe de Golgi. Les virions nouvellement formés sont ensuite transportés à l’intérieur des vésicules de Golgi jusqu’à la surface cellulaire, où ils sont crachés hors de la cellule, explique la virologue et biologiste cellulaire Carolyn Machamer de l’Université Johns Hopkins de Baltimore (Maryland), qui a étudié les coronavirus pendant 30 ans.
Mais en décembre, la biologiste cellulaire Nihal Altan-Bonnet de l’US National Heart, Lung, and Blood Institute et ses collègues, ont signalé qu’ils avaient détecté des coronavirus quittant la cellule à travers des lysosomes — des « poubelles cellulaires » pleines d’enzymes qui se décomposent. Le blocage de la voie sécrétoire basée sur Golgi n’a pas semblé affecter la quantité de virus infectieux libérée, explique Altan-Bonnet. Les preuves de son équipe suggèrent que les protéines virales forment une enveloppe en bourgeonnant dans le RE, puis prennent le relais des lysosomes pour sortir de la cellule. Les chercheurs testent actuellement des inhibiteurs qui bloquent le processus de sortie lysosomale en tant que candidats antiviraux potentiels.
Laisser une cellule à travers le Golgi ou les lysosomes est lent et inefficace par rapport au bourgeonnement d’une membrane plasmique, de sorte que les scientifiques ne savent pas pourquoi le SARS-CoV-2 le fait. Machamer soupçonne que la composition lipidique d’une enveloppe dérivée de Golgi ou de lysosome est en quelque sorte plus bénéfique pour le virus que celle de la membrane plasmique. « Si nous comprenions un peu mieux cette partie, il y aurait de grandes opportunités pour de nouvelles thérapies antivirales », dit-elle.